appels <- 3
peren <- "groen"
appels + peren2 Programmeren in R
2.1 Typen variabelen
We hebben in het eerste onderdeel al een beetje kennis gemaakt met variabelen, door letters bepaalde numerieke waarden te geven (a <- 2, b <- 9.5, etc). Maar je kan in variabelen niet alleen nummers opslaan, maar ook een heleboel andere type gegevens. De meest belangrijke variabelen zijn:
Numeric -
1,2,3, maar ook2.158196zijn numericLogical -
TRUEenFALSE, ook welT/Fof1/0Character (of: string) -
"a","Hoi","ATGCTAT", of"Dit is een zin"
2.2 Geen appels met peren vergelijken
In R kun je berekeningen uitvoeren, maar je kan natuurlijk geen appels met peren vergelijken. Bekijk bijvoorbeeld de volgende code:
Als je de bovenstaande code probeert uit te voeren, geeft R een foutmelding:
Error in appels + peren : non-numeric argument to binary operator1
Het is erg prettig dat R deze foutmelding geeft. Hoewel dit een overduidelijk onzinnige som is, kan het ook zijn dat je met de beste intenties een foutje maakt!
We moeten dus wel dezelfde type dingen bij elkaar optellen. Echter kan je natuurlijk ook niet alles bij elkaar optellen. Als we bijvoorbeeld zowel appels als peren een kleur geven (rood en groen), krijgen we dezelfde error:
appels <- "rood"
peren <- "groen"
appels + perenError in appels + peren : non-numeric argument to binary operator
Je kunt woorden namelijk helemaal niet optellen. Wel zou je natuurlijk de totale lengte van de woorden bij elkaar op kunnen tellen (4+5=9), of de woorden aan elkaar kunnen plakken waardoor we "rood groen" krijgen2.
2.3 Condities
Vaak is het handig om in een script te controleren of aan een bepaalde voorwaarde wordt voldaan. Hiervoor kun je if, else, en else if gebruiken. Ook kunnen we met print() een resultaat weergeven in de console3. Als we deze dingen combineren, krijgen we:
leeftijd_student <- sample(16:19,1) # genereer willekeurige leeftijd tussen 16 en 19
if(leeftijd_student >= 18){
print("Student is volwassen")
} else{
print("Student is niet volwassen")
}Om voorwaarden te controleren, gebruik je zogeheten conditionals, die teruggeven of iets waar (TRUE) of niet waar (FALSE) is. De gekrulde haakjes { en } geven de zogenoemde scope aan. Alles wat tussen deze gekrulde haakjes staat wordt alleen uitgevoerd als aan de conditie wordt voldaan. In het voorbeeld hierboven gebruiken we >= (groter dan of gelijk aan) om te controleren of de student minstens 18 jaar oud is. Veelgebruikte conditionals zijn:
a == b: TRUE als a gelijk is aan ba != b: TRUE als a niet gelijk is aan ba > b: TRUE als a is groter is dan ba < b: TRUE als a is kleiner is dan ba >= b: TRUE als a is groter is dan of gelijk is aan ba <= b: TRUE als a is kleiner is dan of gelijk is aan b
Daarnaast kun je eventueel meerdere condities combineren:
a==b & b!=c: TRUE als a gelijk is aan b én b niet gelijk is aan ca<b | b>c: TRUE als a kleiner is dan b óf b groter is dan c
Als laatste kun je nog veel meer combinaties controleren door if en else in elkaars scope terug te laten komen (dit wordt ook wel het “nesten” van condities genoemd). Zo kunnen we binnen het ene if statement weer andere condities controleren. Als we bijvoorbeeld twee dobbelsteenworpen simuleren, kunnen we precies controleren welke dobbelsteen hoger is dan 4:
d1 <- sample(1:6,1) # dobbelsteen 1
d2 <- sample(1:6,1) # dobbelsteen 2
if (d1 > 4) {
if (d2 > 4) {
print("d1 en d2 zijn hoger dan 4")
} else {
print("d1 is hoger dan 4, maar d2 niet")
}
} else if(d2 > 4) {
print("d1 is niet hoger dan 4, maar d2 wel")
} Let op: als je de bovenstaande code goed leest, zie je dat er helemaal niets wordt geprint als geen van beide dobbelstenen groter dan 4 is.
2.4 Data structuren
Het is vaak heel handig om je variabelen in lijsten of tabellen op te slaan. Dergelijke lijsten of tabellen worden meestal data structures genoemd. De meest belangrijke data structures in R zijn:
Vectors (lijst van elementen van hetzelfde type)
Matrices (tabel van elementen van hetzelfde type)
Dataframes (tabel waarin elke kolom een ander type data kan zijn)
Vectoren
Om een vector te maken, kunnen we deze handmatig intypen:
mijnvector <- c(1,2,0.1,-5)
mijnvector[1] # geeft het eerste getal
mijnvector[2] # geeft het tweede getal, etcMaar handmatig getallen invoeren is niet altijd handig. Gelukkig kun je met R een boel lijstjes op een snellere manier automatisch genereren:
1:4 # 1, 2, 3, 4
5:2 # 5, 4, 3, 2
rep(2,4) # 2, 2, 2, 2
seq(0,4.5,1.5) # 0.0, 1.5, 3.0, 4.5
sample(1:6,4) # 4, 6, 3, 5 (iedere keer anders!)
sample(letters,4) # "f", "j", "o", "y" (iedere keer anders!)
runif(2) # 0.9965360 0.8978062 (iedere keer anders!)De laatste vijf regels van het bovenstaande script zijn voorbeelden van “functies”. Een functie is een herhaalbaar blokje code die je aanroept met de naam (rep, seq, sample), gevolgd door haakjes met daartussen eventuele argumenten. De functie zet deze argumenten om in een zogeheete return-waarde, wat bijvoorbeeld een getal of een lijst getallen is. Je leert meer over functies in het onderdeel Paragraaf 2.8.1.
Je kan met vectoren ook rekenen. Zo kan je bijvoorbeeld twee vectoren bij elkaar optellen, of één en hetzelfde getal bij alle elementen van een vector optellen:
# Rekenen met vectoren
x <- 1:3 # 1, 2, 3
x + 10 # 11 12 13
x + 10 # 11 12 13
y <- c(1, 10, 4) # 1 10 4
x + y # 2 12 7
x * y # 1 20 12
x + y / 100 # 1.01 2.10 3.04
(x + y) / 100 # 0.02 0.12 0.07Let op! Zoals je ziet staat er twee keer x + 10. Hoewel we de eerste keer al 10 bij x hebben opgeteld, is de waarde van x niet aangepast. De tweede keer is het antwoord dus precies hetzelfde, want x is niet veranderd. Als we in plaats hiervan x <- x + 10 hadden gebruikt, zou de waarde van x wel aangepast worden:
x <- x + 10 # x bevat nu 11, 12, 13
x <- x + 10 # x bevat nu 21, 22, 23
x <- x / 10 # x bevat nu 2.1, 2.2, 2.3Let ook op het subtiele verschil tussen de regels x + y / 100 en (x + y) / 100. Net als bij wiskunde gaan haakjes voor delen en vermedigvuldigen, dus het resultaat van deze twee regels is anders. Soms moet je dus goed aangeven in welke volgorde je de berekeninng wil uitvoeren!
Als laatste is het belangrijk om te weten dat spaties in R meestal niets betekenen (dit geldt voor de meeste programmeertalen). Zodoende is x<-x+10 hetzelfde als x <- x + 10, en rep(2,1) hetzelfde als rep(2, 1). Een paar uitzonderingen is dat spaties in het pijltje om variabele toe te kennen niet kunnen (x < - 1 werkt niet). Ook doen spaties in character strings er wel toe, want daar telt spatie als een karakter. Zodoende zijn "appels", " appels" en "appels " niet hetzelfde:
"appels" == "appels" # TRUE
"appels" == "appels " # FALSE
"appels" == " appels" # FALSEDe bovenstaande situatie lijkt misschien een beetje vreemd, maar komt vaak voor omdat er per ongeluk ergens een spatie in je data staat (een spatie valt niet zo snel op!). Het is dus goed om hierop attent te blijven.
Matrices
Matrices in R kun je maken door de functie matrix aan te roepen, die je waarden van de matrix en het aantal rijen als argumenten geeft:
matrix(c(1, 2, 3, 4,5, 6, 7, 8, 9), nrow=3)
matrix(c(1, 2, 3, 4,5, 6, 7, 8, 9), nrow=3, byrow=T)Zoals je ziet zijn er geen comments geplaatst bij deze twee regels. Voer de bovenstaande code zelf uit, en kijk naar de verschillende uitkomsten van de eerste en de tweede regel. Je kan de matrix ook vullen met andere waarden:
kansen_matrix <- matrix(runif(9), nrow=3) # willekeurige getallen tussen 0 en 1
percentage_matrix <- kansen_matrix*100 # de kansen matrix keer 100, dus percentages
letter_matrix <- matrix(sample(letters,9), nrow=3) # willekeurige lettersData frames
De laatste (en voor biologen meest belangrijke) data structuur in R is de data frame. In een dataframe bevat elke kolom een ander gegeven (naam, leeftijd, haarkleur), en is elke rij een apart observatie. Een dergelijke structuur is heel handig om wetenschappelijke data te verzamelen. Ook deze kun je weer handmatig invoeren:
biologen <- data.frame(naam=c("Lina","Yassin","Bram"),
leeftijd=c(19,18,80),
haarkleur=c("Blauw","Groen", "Paars"))
Als je deze dataset wil bekijken in Rstudio, kun je View(biologen) typen. Dit opent een tabblad waarin je de data kan verkennen en sorteren (voor grotere datasets is dit natuurlijk erg handig!).
Het is natuurlijk meestal niet handig om handmatig data frames in te typen in R. In plaats daarvan kun je grote datasets vaak opslaan of downloaden in een groot tekstbestand, bijvoorbeeld in een comma-seperated-value (csv) bestand. Een voorbeeld van de eerste paar regels van een dergelijk csv-bestand, zie je hieronder.
datapunt,week,maand,jaar,positieve_testen
1,1,10,2021,221
2,2,10,2021,271
3,3,10,2021,344
4,4,10,2021,507
5,5,10,2021,514Als je een dergelijk tekstbestand op je computer hebt opgeslagen, kun je deze inlezen met de functie read.table of read.csv:
covid_data <- read.csv("E:/Files/covid.csv") # waar het bestand staat
head(covid_data) # bekijk de eerste 6 rijen
View(covid_data) # bekijk de hele tabel in Rstudio2.5 Indexing
Nu je weet hoe je tabellen in kan lezen, is het belangrijk om te leren over “indexing”. Indexing is het selecteren van specifieke waarden uit een vector, matrix, of dataframe. Dit doe je door de positie (index) van deze elementen tussen vierkante haakjes aan te geven:
jaartallen <- 2009:2024 # Jaren dat ik biologie "studeer"
jaartallen[6:9] # 2014 2015 2016 2017
m <- matrix(runif(9),nrow=3) # Willekeurige getallen in een matrix
m[1,2] # rij 1, kolom 2
m[1,] # rij 1 volledig
m[,2] # kolom 2 volledig
m[1:2,3] # van rij 1 t/m 2, alleen de 3e kolom
biologen <- data.frame(naam=c("Lina","Yassin","Bram"),
leeftijd=c(19,18,80),
haarkleur=c("Blauw","Groen", "Paars"))
biologen[1,2] # eerste rij, tweede kolom (leeftijd: 19)Omdat in data frames de kolommen namen hebben, kun je ook deze gebruiken om specifieke datapunten te selecteren:
biologen[1,"leeftijd"] #identiek aan biologen[1,2], maar het is wat duidelijker wat je selecteertJe kunt met dataframes ook met het dollar-teken een gehele kolom opvragen, een kolom bewerken, of een kolom toevoegen:
biologen$leeftijd
biologen$leeftijd <- biologen$leeftijd + 1 # De leeftijd van alle biologen wordt met 1 verhoogd
biologen$rol <- c("student","assistent","docent") # Een nieuwe kolom genaamd 'rol'2.6 Selecteren uit een dataframe
Je kunt ook bovengenoemde conditionals (groter dan, gelijk aan, etc.) gebruiken op vectoren. Dit geeft dan een vector van dezelfde lengte, met voor elke positie of aan de conditie voldaan wordt. Als we bijvoorbeeld de zelf-gemaakte biologen-dataframe nog eens bekijken, dan kunnen we een vector maken die voor de leeftijd-kolom zegt wie er jonger is dan 20:
biologen <- data.frame(naam=c("Lina","Yassin","Bram"),
leeftijd=c(19,18,80),
haarkleur=c("Blauw","Groen", "Paars"))
jong <- biologen$leeftijd < 20 # geeft TRUE TRUE FALSEMet deze vector kunnen we dan een selectie maken uit een dataframe. Weet je nog dat we hierboven zeiden dat je uit dataframe kan selecteren door [rijen,kolommen] aan te geven? Met onze nieuwe jong-vector, kunnen we dus de juiste rijen pakken:
biologen[jong,] # alleen biologen jonger dan 20Dit is natuurlijk vooral handig als je een grotere dataset hebt, zoals de bovengenoemde covid-data. Met een dergelijke dataset is het niet handig om handmatig aan te geven welke rijen je wil behouden!
2.7 Files, paden, en de ‘working directory’
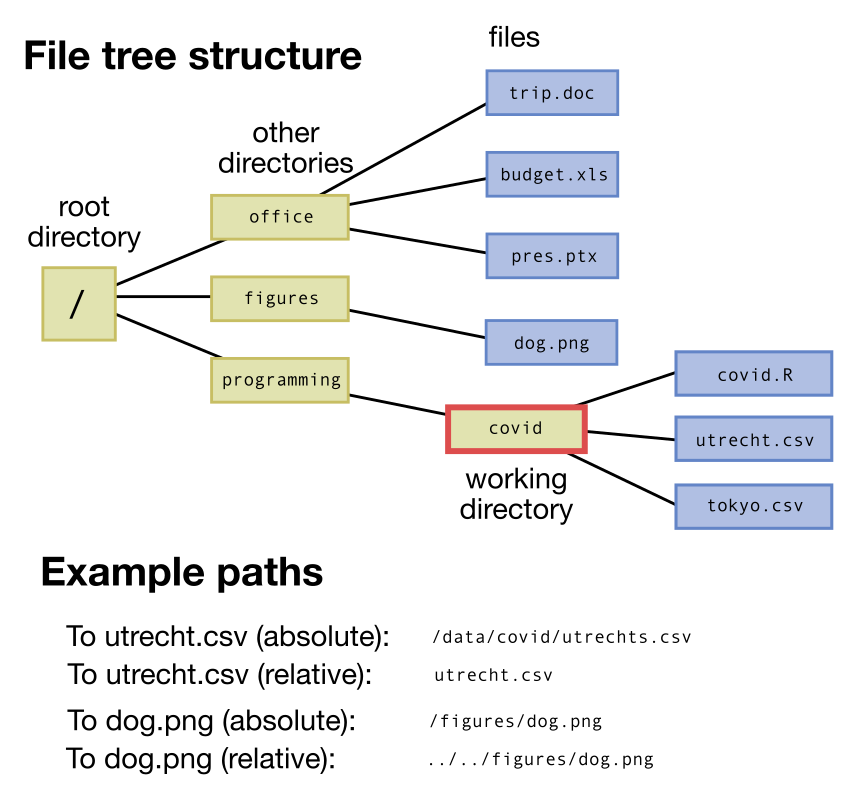
Om (zoals hierboven) een bestand in te kunnen lezen, moet Rstudio wel weten waar het bestand staat. Op computers staan alle bestanden in een hierachische structuur (zie bijvoorbeeld onderstaande Figuur). Hoewel je misschien gewend bent om door deze structuur van “mappen” te klikken met de muis, moet je in een script altijd precies vertellen waar het bestand staat. Standaard zoekt Rstudio vaak in de home directory van je besturingsysteem (/Users/Naam in MacOs, C:/Users/UserName/Documents in Windows). De directory vanuit waar Rstudio naar het bestand zoekt, heet de Working Directory (rood omlijnd in de figuur). Door de Working Directory goed in te stellen, hoef je niet elke keer een heel lang pad (dit heet het absolute pad) naar het bestand in te typen, maar kun je met een korter pad omschrijven waar het bestand staat (dit heet het relatieve pad). Vaak is het dus handig om je Working Directory aan te passen naar dezelfde map waar je het script hebt opgeslagen, zodat je alle data, scripts, en grafieken een beetje bij elkaar kan houden. Dit kan je doen door, nadat je je script ergens hebt opgeslagen, te navigeren naar Session > Set Working Directory > To Source File Location, of door rechts te klikken op je script-naam bovenin het script-venster, en Set Working Directory te kiezen.
2.8 Functies
Voorbeelden van functies en ‘help’
In het onderdeel Paragraaf 2.4.1 heb je al een aantal voorbeelden gezien van functies, herhaalbare blokjes code die je gemakkelijk kan aanroepen. Functies hebben een input (waarde(n) die je meegeeft als zogeheten argumenten), en output (waarde(n) die het terug geeft, de return-waarde). Er bestaan standaard in R een heleboel functies, waarvan je een aantal in deze cursus nog veel zal tegenkomen:
read.csv, leest comma-separated-values bestand in als dataframeprintofcat, geeft iets (variabelen of tekst) weer in de consoleplot, om data te plotten in een grafieklengthgeeft de lengte van een vectormean, gemiddelde van een vectorsd, standaarddeviatie van een vectorsum, som van een vectormingeeft de kleinste waarde in een vectormaxgeeft de grootste waarde in een vectorsort, sorteert een vector
Als je graag meer wil weten over hoe een functie werkt, kun je de naam van de functie typen met een ? ervoor:
?meanDit geeft extra uitleg over deze functie in het “Help” venster (rechtsonderin Figuur 1.2).
Zelf functies maken
Je kan ook zelf functies maken. Dit is handig als je meerdere keren dezelfde specifieke berekening wil uitvoeren. Je moet hiervoor aangeven wat de argumenten zijn, welke berekening op basis van deze argumenten wordt uitgevoerd, en wat er door de functie wordt teruggegeven (de return-waarde). Hier is een voorbeeld:
# Definitie van de functie
som_kwadraten <- function(a, b) {
resultaat <- a^2 + b^2
return(resultaat)
}
# Voorbeeld gebruik van de functie
som_kwadraten(1,2) # 5
som_kwadraten(3, 4) # 25
som_kwadraten(0.5, 0.1) # 0.26Bij het definiëren van de functie geven de gekrulde haken aan wat de zogeheten scope van de functie is: dit blokje code behoort alleen deze functie toe. Je ziet deze scopes ook terug wanneer je op bepaalde voorwaarden gaat toetsen, als je ‘loops’ gaat gebruiken, wat in het volgende deel uitgelegd wordt.
2.9 Loops
Hoewel functies het makkelijker maken om dezelfde berekening meerdere keren uit te voeren, wat nu als je een functie 100 keer wil aanroepen? Hebben we dan 100 regels nodig? Het antwoord is nee, want elke programmeertaal (dus ook R) heeft manieren om stukken code gemakkelijk te herhalen. In R bestaan veel verschillende manieren om dit te doen (repeat, apply, while), maar in deze introductie maken we vooral kennis met de meest-gebruikte loop in de programmeer-wereld: de for-loop.
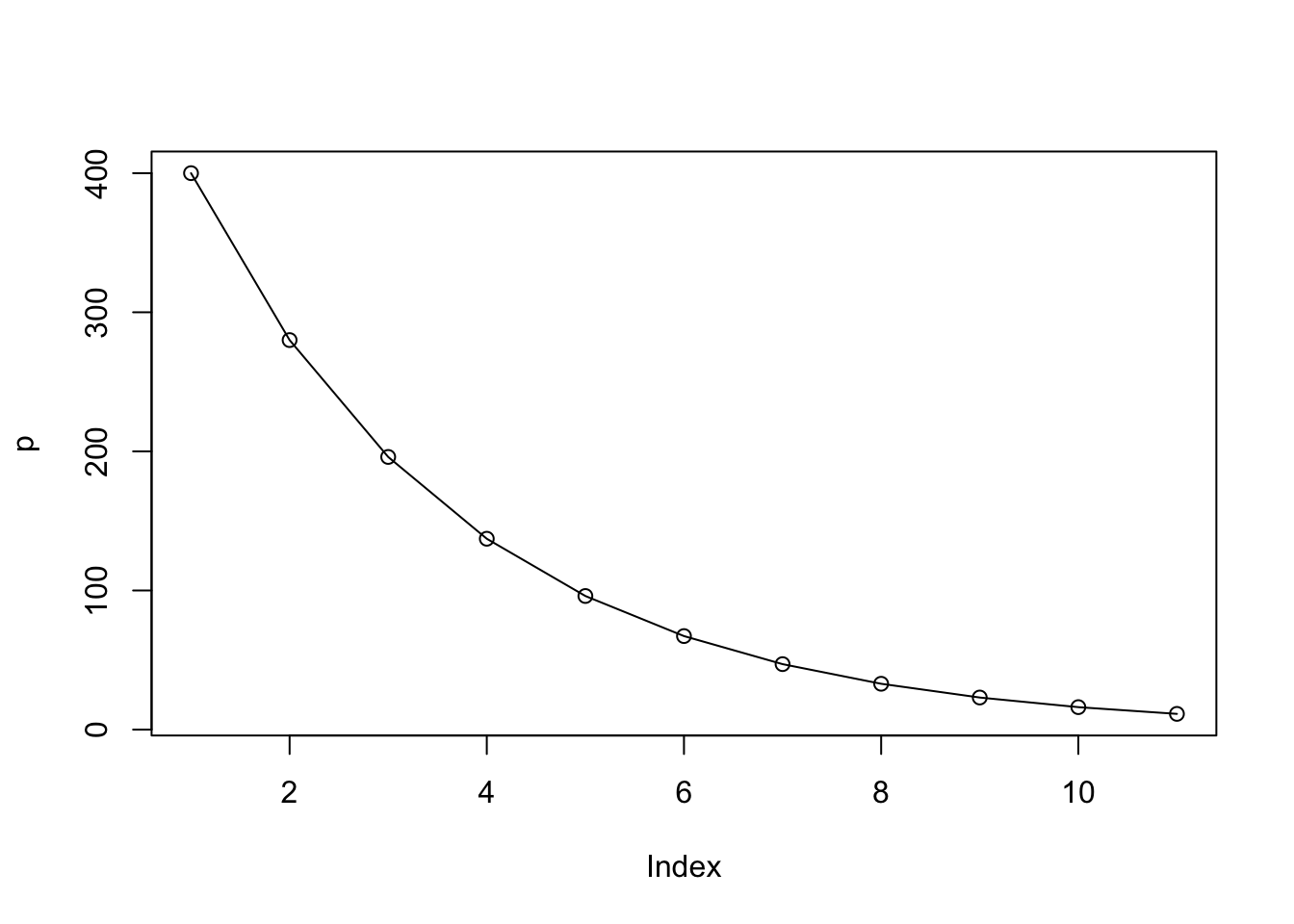
Laten we bijvoorbeeld de exponentiële afname van paracetamol in het bloed beschrijven met een simpele “simulatie”. Iemand neemt 400 milligram paracetamol in, wat ieder uur (ongeveer) met 70% afneemt:
p <- 400 # hoeveelheid paracetamol
d <- 0.7 # afname (d van decay)
# Herhaal iets 10 keer, waarbij 'i' steeds met 1 toeneemt
for(i in 1:10){
p <- p*d
}
cat('Na 10 uur is er',p,'milligram paracetamol in het bloed') # 11.299 mgAan het einde van deze for-loop kunnen we kijken wat de waarde van p is door in de console p te typen.
> p
[1] 11.29901Zoals we eerder hadden besproken, staat de [1] voor het feit dat dit de eerste en enige waarde is die in de variable p is opgeslagen. We hebben nu dus alleen de laatste waarde bewaard. Wellicht willen we echter graag bijhouden hoeveel paracetamol er in elke stap aanwezig was, zodat we dat na onze loop kunnen plotten. Om dit te doen, kunnen we de i als index gebruiken om een vector te vullen:
p <- 400 # hoeveelheid paracetamol
d <- 0.7 # afname (d van decay)
# Herhaal iets 10 keer, waarbij 'i' steeds met 1 toeneemt
for(i in 1:10){
p[i+1] <- p[i]*d
}
plot(p, type='o')Omdat dit voorbeeld om simpele exponentiele afname gaat, hadden we de bovenstaande berekening ook “in één keer” kunnen uitrekenen door \(p \cdot d^{10}\) uit te rekenen. Laten we dat even controleren:
p <- 400 # hoeveelheid paracetamol
d <- 0.7 # afname (d van decay)
p*d^10 # 11.299 mg, dat klopt dus!Dit antwoord komt inderdaad overeen met onze berekening met de for-loop. Hoewel deze loop dus misschien wat onnodig lijkt, hebben we niet altijd zo’n simpele formule beschikbaar. Bovendien willen we wellicht iets veel specifiekers simuleren, bijvoorbeeld dat de persoon gedurende een periode van 100 uur elke 10 uur opnieuw paracetamol inneemt:
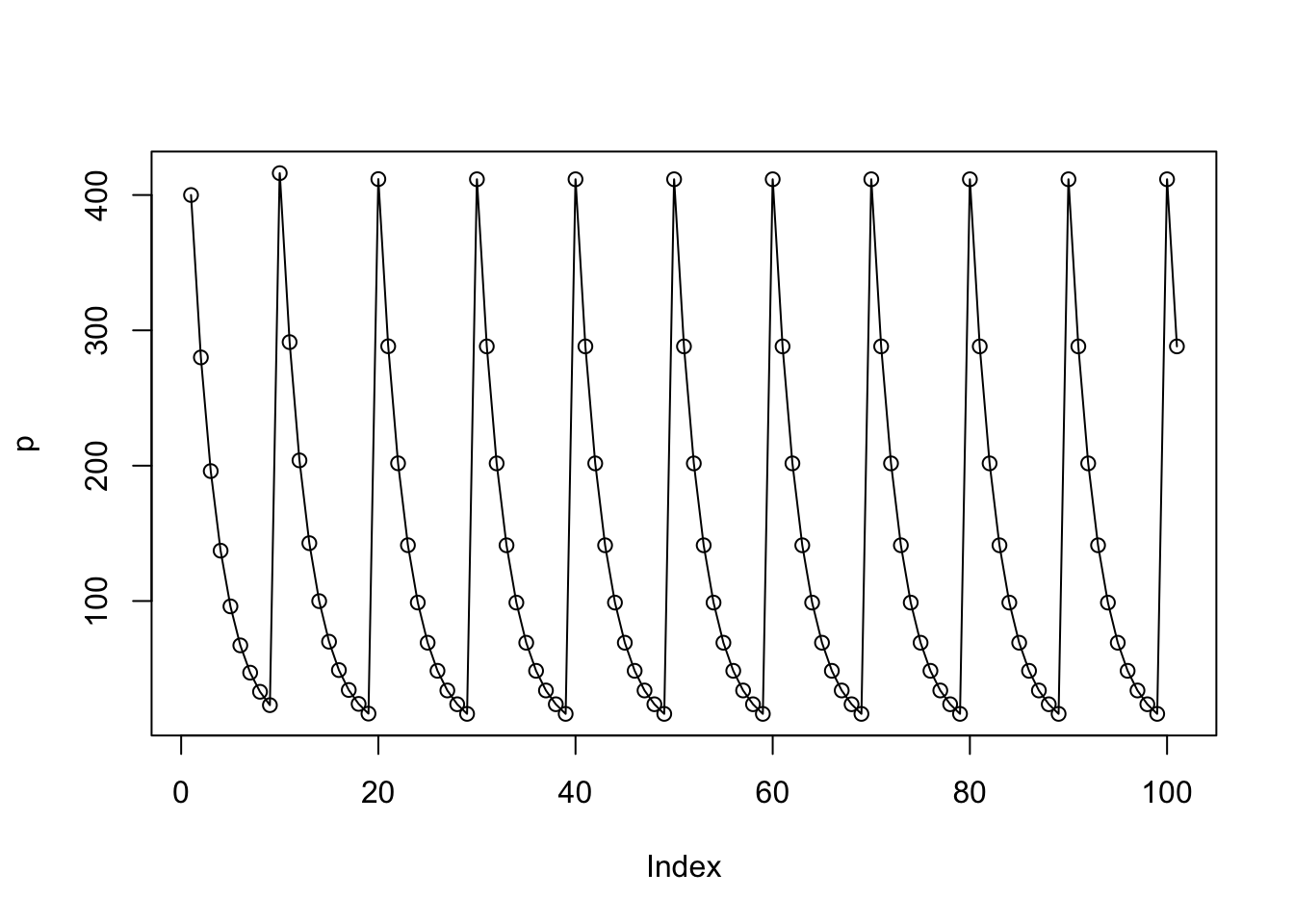
p <- 400 # hoeveelheid paracetamol
d <- 0.7 # afname (d van decay)
for(i in 1:100){
if(i%%10==0){ # is i precies deelbaar door 10?
p[i] <- p[i] + 400
}
p[i+1] <- p[i]*d
}
plot(p, type='o')Of dat de persoon met 10% kans elk uur een paracetamol inneemt:
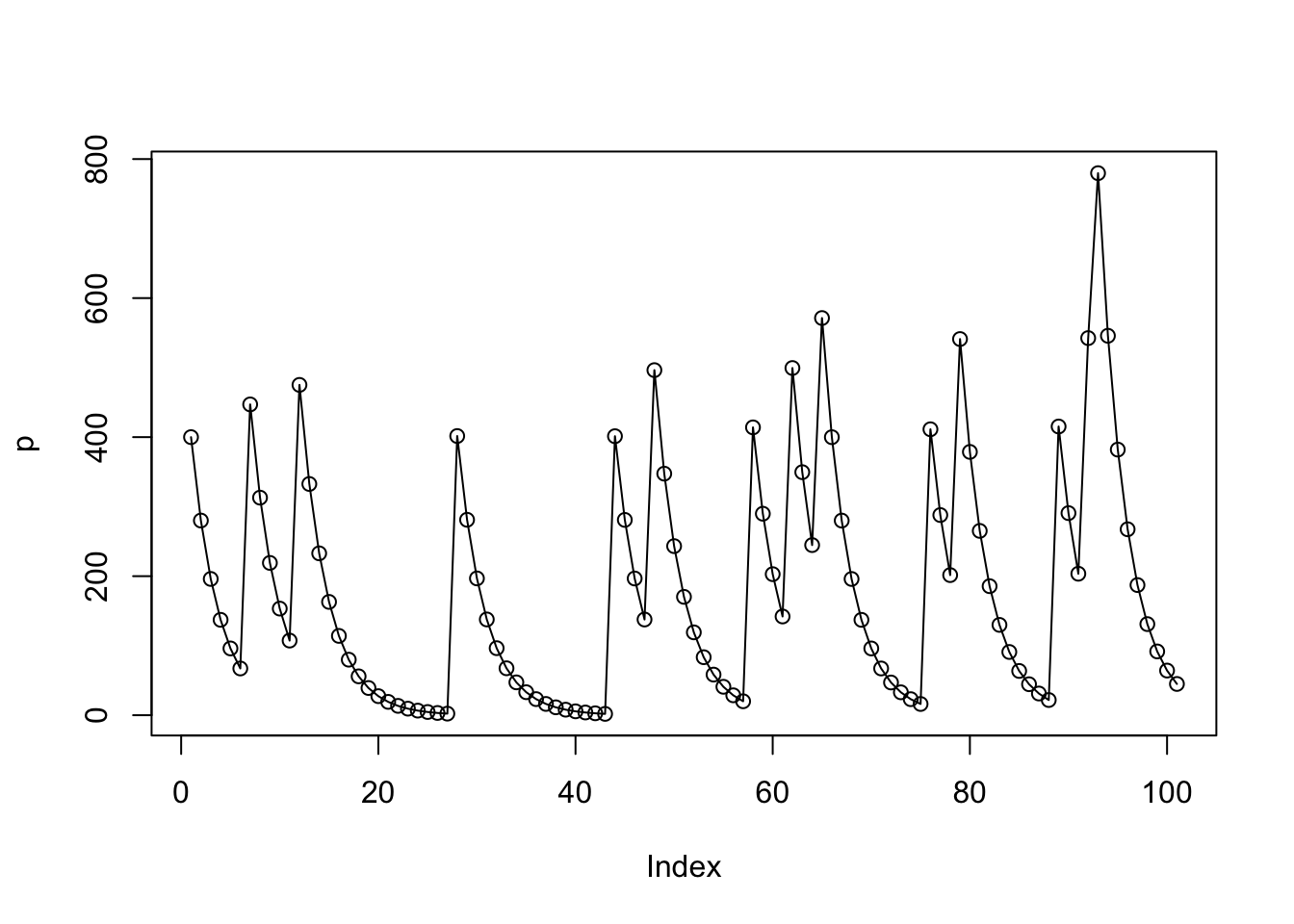
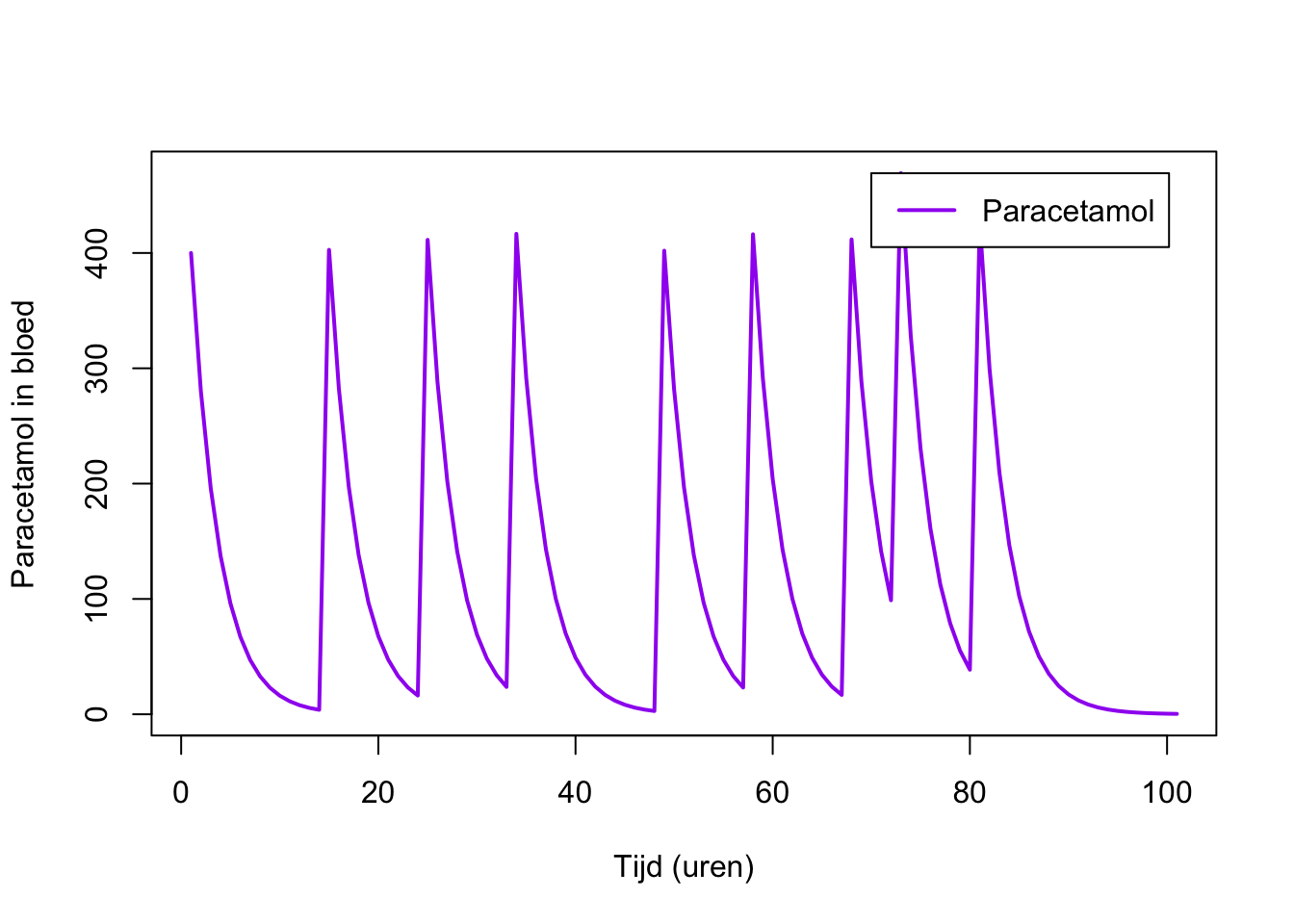
2.10 Grafieken plotten met R
Om de bovenstaande plot van de paracetamol afname iets vollediger en mooier te maken, kunnen we meer argumenten aan de plotfunctie toevoegen:
p <- 400 # hoeveelheid paracetamol
d <- 0.7 # afname (d van decay)
for(i in 1:100){
if(runif(1)<0.1){ # is willekeurig getal (tussen 0 en 1) kleiner dan 0.1?
p[i] <- p[i] + 400
}
p[i+1] <- p[i]*d
}
plot(p, type='l', lwd=2, col="purple", xlab="Tijd (uren)", ylab="Paracetamol in bloed")
legend(70, max(p), legend=c("Paracetamol"), col=c("purple"),lty=1,lwd=2)Er zijn binnen R nog veel meer manieren om data te plotten, zoals bijvoorbeeld de boxplot die je bij het onderdeel statistiek zeker nog tegen zal komen. Daarnaast zijn er in R veel packages beschikbaar die kunnen helpen bij het maken van mooie grafieken, zoals ggplot, maar daar gaan we in deze cursus niet dieper op in.
2.11 Opgaven
Oefening 2.1 (Packages)
R wordt wereldwijd door veel biologen gebruikt. Daarom kun je vaak online veel hulp vinden als je een bepaalde analyse wil uitvoeren met R. Er zijn bijvoorbeeld heel veel packages en Rscripts beschikbaar om data te visualiseren, fylogenetische bomen te analyseren, diversiteitsanalyses uit te voeren, etc.. Een package moet je één keer installeren, en kan je daarna eenvoudig inladen.
a. Voer in R de functie rtree(10) uit. Wat is je resultaat?
We installeren het package ‘ape’ met de onderstaande code en laden deze in (installeren hoeft maar één keer, inladen moet iedere keer dat je Rstudio opnieuw opstart).
install.packages("ape") # Installatie. Hoeft maar 1 keer!
library(ape) # inladen van het packageb. Voer nogmaals rtree(10) uit. Wat is het resultaat?
We kunnen de uitkomst uit vraag b ook plotten met R:
# installeren hoeft niet meer :)
library(ape) # inladen
tree <- rtree(10)
plot(tree)c. Voer het script een aantal keren uit. Is het resultaat iedere keer hetzelfde? Waar denk je dat de ‘r’ in rtree voor staat?
d. Bestudeer wat het argument 10 doet.
e. Bestudeer welke andere argumenten deze functie heeft.
Naast packages kan je ook een Rscript inlezen. Dit doet je met de functie source(), waar je tussen haakjes aangeeft waar het script te vinden is (dit kan zowel op je computer staan als online). Zo kan je bijvoorbeeld een eerder-gebruikt script in je nieuwe script inladen, of een groot script geschreven door iemand inlezen. Later in de cursus ga je bijvoorbeeld het Grind.R script inlezen vanaf de website van Rob de Boer, maar hier komen we later in de cursus op terug.
Oefening 2.2 (Pinguïns)
Sommige packages zijn er vooral om voorbeeld-datasets te verkrijgen. Een daarvan is palmerpenguins:
install.packages("palmerpenguins")
library(palmerpenguins)
head(penguins)Voer de code hierboven uit. Je hebt nu de dataframe penguins tot je beschikking.
a. Hoeveel soorten pinguïns staan er in de data set?
De kolom penguins$flipper_length_mm geeft de flipper lengtes van de pinguïns. Echter is er bij enkele metingen iets misgegaan, waardoor deze de waarde NA (Not Available) heeft.
b. De functies min, max en mean geven respectievelijk de kleinste, grootste, en gemiddelde waarde van een lijst. Bereken deze waarden voor de flipper lengtes (hint: je kunt deze functies een tweede argument na.rm=TRUE meegeven om de NA’s te verwijderen).
c. Gebruik de functie summary om te berekenen voor welk percentage van de pinguïns de sekse niet is bepaald.
Oefening 2.3 (Bacteriegroei)
Een bioloog heeft een experiment uitgevoerd waarin hij de groeisnelheid van bacteriën heeft gemeten bij verschillende temperaturen. De data is als volgt:
- Temperatuur: 20, 25, 30, 35, 40 graden Celcius
- Groeisnelheid: 2.0, 2.25, 2.65, 2.35, 2.2 per uur
- Stel een dataframe samen met twee kolommen, één voor temperatuur en één voor groeisnelheid
- Bepaal bij welke temperatuur de groeisnelheid maximaal is (tip: gebruik de functie
which.max()om de index met de hoogste waarde te vinden)
Een groeisnelheid van 2 betekent dat de bacteriën elk uur verdubbelen. Na 2 uur zijn er dus 4 keer zoveel bacteriën, na 3 uur 8 keer, en uiteindelijk na tien uur 1024 keer meer.
Bepaal voor elke temperatuur hoeveel keer meer bacteriën er zijn na 10 uur. Voeg dit toe als nieuwe kolom ‘totaalgroei’ in je dataframe.
Controlleer met R of de totaalgroei met een groeisnelheid van 2 inderdaad uitkomt op 1024.
Met plot() kun je de waarden van twee kolommen tegen elkaar plotten. Als eerste eerste argument geef je de waarden op de x-as, en als tweede de waarden op de y-as.
- Plot de groeisnelheid tegen de temperatuur. Geef je grafiek een kleur, een titel, en label de assen.
Oefening 2.4 (Vissen vrijlaten)
Een ecoloog heeft data verzameld over de lengte van vissen in een vijver, omdat hij vissen langer dan 15 cm wil uitzetten in een natuurlijk meer. De data staat in de volgende vector:
# De dataset met lengtes van vissen (in cm):
lengtes <- c(12, 18, 7, 21, 15, 16, 14, 10, 20, 17)Door een vector te controleren op een bepaalde voorwaarde krijg je een nieuwe vector terug, met voor elke meting of dit waar is of niet. Omdat TRUE gelijk staat aan 1, en FALSE gelijk staat aan 0, kun je met de functie sum vervolgens bepalen hoeveel metingen aan een bepaalde voorwaarde voldoen.
a. Gebruik de bovenstaande informatie om de tellen hoeveel vissen groter of gelijk aan 15 cm zijn.
b. Bereken het percentage vissen dat te klein zijn om uit te zetten.
In plaats van specifieke waardes als index te gebruiken, kun je ook een TRUE/FALSE-vector gebruiken om data uit een lijst te selecteren.
c. Selecteer alle lengtes van vissen die groot genoeg zijn (>=15 cm).
d. Gebruik een for-loop om vissen stuk voor stuk te beoordelen, en print voor elke vis of ze groot genoeg zijn.