library(NHANES)
data(NHANES)
sum(NHANES$Gender == "female")[1] 5020categorisch & nominaal
numeriek & discreet
categorisch & ordinaal
numeriek & continu
numeriek & continu
discreet & numeriek
–
–
MaritalStatus is een nominale variabele met niveaus “Divorced”, “LivePartner”, “Married”, “NeverMarried”, “Separated”, of “Widowed”.
Height is een numerieke, continue variabele. De eenheid is cm.
nBabies is een numerieke, discrete variabele. Bij mannen is NA ingevuld, voor Not Available. Dit is de nette manier om in R missende gegevens aan te geven.
Er zijn 5020 vrouwen in de dataset:
library(NHANES)
data(NHANES)
sum(NHANES$Gender == "female")[1] 5020Bij een klein aantal vrouwen is 0 ingevuld:
sum(NHANES$nBabies == 0, na.rm = TRUE)[1] 7Dat zijn er wel erg weinig voor zo’n grote groep. Het vermoeden is dus dat bij vrouwen zonder kinderen ook vaak NA is ingevuld.
De eerste regel van de uitvoer is:
Classes ‘tbl_df’, ‘tbl’ and 'data.frame': 10000 obs. of 76 variables:We lezen hier af dat er 10000 waarnemingen zijn van 76 variabelen.
| a. | b. | c. | d. | e. | f. | |
|---|---|---|---|---|---|---|
| Symmetrisch | \(\checkmark\) | \(\checkmark\) | \(\checkmark\) | \(\checkmark\) | \(\checkmark\) | |
| Links-scheef | ||||||
| Rechts-scheef | \(\checkmark\) | |||||
| Klokvormig | \(\checkmark\) | |||||
| Uniform | \(\checkmark\) | |||||
| Bimodaal | \(\checkmark\) |
Ongeveer 54%.
Ongeveer 81%.
Ongeveer 86% weegt hoogstens 100kg, en 54% weegt hoogstens 75kg. Tussen die grenzen zit dus \(86\% - 54\% = 32\%\).
Ongeveer 25kg.
Het gemiddelde gaat nu van een 6,6 naar een 7,8.
De mediaan blijft hetzelfde. De “middelste” waarde van de dataset is namelijk niet veranderd.
Het gemiddelde is een stuk gevoeliger voor uitbijters. In dit geval had het verschuiven van de uitbijter geen enkel effect op de mediaan.
a. en b. Een overzicht van de kwartielen:
\[ 12\ 15\ 18\ 20 \underset{\mathrm{Q1} = 20{,}5}{|} 21\ 23\ 25\ 28 \underset{\mathrm{Q2} = 29}{|} 30\ 32\ 35\ 38 \underset{\mathrm{Q3} = 39}{|} 40\ 42\ 45\ 46 \] De mediaan is Q2 = 29.
\(\text{IKA} = \text{Q3} - \text{Q1} = 39 - 20{,}5 = 18{,}5\).
Nu worden de kwartielen:
\[ 12\ 15\ 18\ 19\underset{\mathrm{Q1} = 19{,}5}{|} 20 \ 21\ 23\ 25\quad \underset{\mathrm{Q2} = 28}{28\phantom{|}} \quad 30\ 32\ 35\ 38 \underset{\mathrm{Q3} = 39}{|} 40\ 42\ 45\ 46 \] De IKA is dus: \[\text{IKA} = \text{Q3} - \text{Q1} = 39 - 19{,}5 = 19{,}5. \]
Let op! In de berekeningen hieronder is het heel belangrijk dat je niet tussendoor afrondt. Als je een eerder resultaat later hergebruikt, gebruik dan het onafgeronde resultaat.
We tellen alle gewichten bij elkaar op: \[\sum_{i=1}^3 X_i = 3{,}0 \textrm{\,kg} + 3{,}4\textrm{\,kg} + 4{,}2\textrm{\,kg} = 10{,}6\textrm{\,kg}.\]
Boven de deelstreep worden alle getallen bij elkaar opgeteld, eronder staat het aantal getallen, namelijk 3. Dit is dezelfde formule als die van het gemiddelde. Het resultaat is:
\[\overline{X} = \frac{\sum_{i=1}^3 X_i}{3} = 3{,}53\textrm{\,kg}.\]
\(i=2\) onder het sommatieteken geeft aan dat we beginnen met optellen bij het tweede datapunt. We tellen dus alleen \(X_2\) en \(X_3\) bij elkaar op:
\[\sum_{i=2}^3 X_i = 3{,}4\textrm{\,kg} + 4{,}2\textrm{\,kg}=7{,}6\textrm{\,kg}.\]
Er staat nu niet 3 bovenin het sommatieteken, maar \(n\). Maar \(n\) is hier gelijk aan 3, en dus we krijgen hier hetzelfde antwoord als bij a: \(10{,}6\textrm{\,kg}\).
Omdat het kwadraat-teken hier binnen de sommatie staat moeten we elk datapunt eerst kwadrateren, en daarna de resultaten bij elkaar optellen:
\[\sum^n_{i=1} X_i^2 = (3{,}0\textrm{\,kg})^2 + (3{,}4\textrm{\,kg})^2 + (4{,}2\textrm{\,kg})^2 = 38{,}2\textrm{\,kg}^2\]
Nu staat de hele sommatie tussen haken en het kwadraat daarbuiten. Tussen haken staat dus het resultaat van a, en dat moeten we kwadrateren:
\[\left( \sum^n_{i=1} X_i\right)^2 = (10{,}6\textrm{\,kg})^2 = 112\textrm{\,kg}^2.\]
We tellen hier de afwijkingen van het gemiddelde (residuen) bij elkaar op. Deze som is altijd gelijk aan nul! Je kunt dat ook wiskundig aantonen: \[ \sum_{i = 1}^n (X_i - \overline{X}) = \left(\sum_{i = 1}^n X_i\right) - \sum_{i = 1}^n \overline{X} = n \overline{X} - n \overline{X} = 0.\]
Hier moeten we eerst het kwadraat van elk verschil uitrekenen. Hierdoor wordt het uiteindelijke resultaat niet meer 0 omdat de negatieve residuen door het kwadrateren positief worden:
\[\sum^n_{i=1} (X_i - \overline{X})^2 = (3{,}0\textrm{\,kg} - 3{,}53\textrm{\,kg})^2 + (3{,}4\textrm{\,kg} - 3{,}53\textrm{\,kg})^2 + (4{,}2\textrm{\,kg}-3{,}53\textrm{\,kg})^2 = 0{,}747\textrm{\,kg}^2.\]
De formule voor de variantie is: \[V_X = \frac{\sum_i^n \left(x_i - \overline{x}\right)^2}{n-1}.\] De teller in deze formule komt overeen met ons antwoord van h; deze hoeven we dus niet nog een keer uit te rekenen. We krijgen dus:
\[V_X = \frac{0{,}747}{3-1} = 0{,}373\textrm{\,kg}^2.\]
Voor de standaarddeviatie nemen we hier de wortel van:
\[s_X = \sqrt{V_X} = 0{,}611\textrm{\,kg}.\]
Standaarddeviatie: 1,0. IKA: 1,3.
Standaarddeviatie: 2,8. IKA: 3,5.
Standaarddeviatie: 0,3. IKA: 0,5.
Standaarddeviatie: 1,8. IKA: 3,1.
Standaarddeviatie: 2,7. IKA: 4,7.
Standaarddeviatie: 0,2. IKA: 0,3.
We kunnen niet de gewone formule voor het gemiddelde gebruiken, want ieder cijfer heeft nu een ander “gewicht”. We kunnen het gewogen gemiddelde berekenen door elk cijfer te vermenigvuldigen met het bijbehorende gewicht, en het resultaat te delen door de som van de gewichten:
\[\frac{10\times 5+60\times 8 + 30\times 7}{10 + 60 + 30}=\frac{10\times 5+60\times 8 + 30\times 7}{100}=7{,}4.\] Het is dus net alsof je 10 keer een 5 hebt gehaald, 60 keer een 8, en 30 keer een 7, met in totaal 100 cijfers.
We kunnen hetzelfde resultaat krijgen als we elk cijfer vermenigvuldigen met het kommagetal dat hoort bij het percentage, want:
\[ \begin{align} \frac{10\times 5+60\times 8 + 30\times 7}{100} &= 0{,}1\times 5+0{,}6\times 8 + 0{,}3\times 7\\ & = 7{,}4. \end{align} \tag{1}\]
De waarnemingen zijn 5, 3, 6, 2, 4, 3, 3, 2, 2, 5. Het gemiddelde is \(\left(\sum_{i = 1}^{10} x_i\right)/10 = 3{,}5\).
We kunnen \(5 + 3 + 6 + 2 + 4 + 3 + 3 + 2 + 2 + 5\) schrijven als \(3 \times 2 + 3\times 3 + 1 \times 4 + 2 \times 5 + 1 \times 6\). Daarom geldt ook: \[ \overline{x} = \frac{\sum_{i = 1}^{10} x_i}{10} = \frac{3\times {\color{darkorange}2} + 3\times {\color{darkorange}3} + 1\times {\color{darkorange}4} + 2\times {\color{darkorange}5} + 1 \times {\color{darkorange}6} }{10}. \tag{2}\]
Het tweede (oranje) getal in de vermenigvuldigingen is dus telkens waarde van de variabele (het aantal vogels), het eerste getal de absolute frequentie van die waarde.
De noemer van Vergelijking 2 kunnen we verrekenen met de zwarte getallen in de teller:
\[ \begin{align} \frac{\sum_{i = 1}^{10} x_i}{10} & = \frac{3\times {\color{darkorange}2} + 3\times {\color{darkorange}3} + 1\times {\color{darkorange}4} + 2\times {\color{darkorange}5} + 1 \times {\color{darkorange}6} }{10}\\ & = \left(\frac{3}{10}\right)\times {\color{darkorange}2} + \left(\frac{3}{10}\right)\times {\color{darkorange}3} + \left(\frac{1}{10}\right)\times {\color{darkorange}4} + \left(\frac{2}{10}\right)\times {\color{darkorange}5} +\left(\frac{1}{10}\right)\times {\color{darkorange}6}\\ & = 0{,}3 \times {\color{darkorange}2} + 0{,}3\times {\color{darkorange}3} + 0{,}1 \times {\color{darkorange}4} + 0{,}2\times {\color{darkorange}5} +0{,}1\times {\color{darkorange}6}. \end{align} \tag{3}\]
Het gevolg is dat we uiteindelijk steeds de relatieve frequentie met de waarde van de variabele vermenigvuldigen.
Vergelijking 3 is niets anders dan de som over alle mogelijke uitkomsten, steeds vermenigvuldigd met hun relatieve frequentie. Dat is precies wat vergelijking Vergelijking 20.7 uitdrukt.
Vergelijk Vergelijking 3 met Vergelijking 1. De boodschap is: als een frequentietabel gegeven is, kun je het gemiddelde uitrekenen door een gewogen gemiddelde uit te rekenen met de relatieve frequenties als de gewichten.
Vogelsoort is een nominale variabele. Een histogram, cumulatieve frequentiepolygoon, of boxplot is dus niet van toepassing. Dan blijft antwoord D, het staafdiagram, over. Dat past ook: een staafdiagram kan voor iedere vogelsoort de frequentie laten zien.
Aangezien we vooral geïnteresseerd zijn in de relatieve frequenties van de zetelverdeling, zou een taartdiagram goed werken.
Het is leuk dat deze visualisatie de zetelverdeling laat zien door letterlijk de opstelling van de zetels (stoelen) in de Tweede Kamer weer te geven. Een nadeel is dat het aantal zetels per partij niet direct in het oog springt; je moet een beetje zoeken naar de zetels van iedere kleur. Een subtieler punt: de zetels zijn niet allemaal even groot weergegeven! Het zetelaantal van een partij en de totale oppervlakte van de bijbehorende kleur komen daardoor niet precies overeen, wat misleidend kan zijn.
naam: nominaal
level: discreet
totale_speeltijd: continu
klassement: ordinaal
console: nominaal.
naam: character
level: numerical
totale_speeltijd: numerical
klassement: character
console: character.
De variabelen klassement en console zijn categorisch en kunnen dus goed als factor worden opgeslagen. Dat geldt op zich ook voor variabele naam, maar omdat de namen uniek zijn wordt de lijst met niveaus dan even lang als de oorspronkelijke vector; deze variabele zal als factor dus niet efficiënter kunnen worden opgeslagen dan character string.
Je kunt klassement direct als factor definiëren:
naam <- c("Mario", "Luigi", "Peach", "Toad", "Bowser")
level <- c(8, 1, 3, 2, 10)
totale_speeltijd <- c(57884, 14841, 7709, 5513, 109867)
# definieer klassement direct als factor:
klassement <- factor(c("Goud", "Brons", "Zilver", "Zilver", "Goud"))
console <- c("Switch", "Switch", "Wii", "Wii", "Switch")
df = data.frame(naam, level, totale_speeltijd, klassement, console)Alternatief is om de kolom eerst als character vector te definieren en achteraf om te zetten naar een factor:
naam <- c("Mario", "Luigi", "Peach", "Toad", "Bowser")
level <- c(8, 1, 3, 2, 10)
totale_speeltijd <- c(57884, 14841, 7709, 5513, 109867)
# definieer klassement eerst als vector van type character:
klassement <- c("Goud", "Brons", "Zilver", "Zilver", "Goud")
console <- c("Switch", "Switch", "Wii", "Wii", "Switch")
df = data.frame(naam, level, totale_speeltijd, klassement, console)
# kolom klassement omzetten naar factor
df$klassement = factor(df$klassement)Vraag de niveaus op:
levels(klassement)[1] "Brons" "Goud" "Zilver"Het is een ordinale variabele. Deze is als een factor opgeslagen, met 5 niveaus:
str(gezondheid) Factor w/ 5 levels "Excellent","Vgood",..: 3 3 3 NA 3 NA NA 2 2 2 ...levels(gezondheid)[1] "Excellent" "Vgood" "Good" "Fair" "Poor" Het valt op dat er veel regels zijn waar niet een van de niveaus maar NA is ingevuld.
De functie na.omit() verwijdert alle NA’s uit de vector.
Voorbeeld:
voorbeeld_vector <- c("Biologie van Dieren", NA, "Microbiologie")
na.omit(voorbeeld_vector)[1] "Biologie van Dieren" "Microbiologie"
attr(,"na.action")
[1] 2
attr(,"class")
[1] "omit"Het resultaat is:
gezondheid.tgezondheid
Excellent Vgood Good Fair Poor
878 2508 2956 1010 187 De functie table() heeft voor ieder niveau van gezondheid geteld hoe vaak die waarde voorkomt in gezondheid. Het resultaat is een absolute frequentietabel; de waardes komen overeen met de tabel uit Tabel 20.2.
Je uiteindelijke code zou er zo uit kunnen zien:
library(NHANES)
gezondheid <- NHANES$HealthGen
gezondheid <- gezondheid[!is.na(gezondheid)]
gezondheid.t <- table(gezondheid)
(gezondheid.f <- gezondheid.t/sum(gezondheid.t))gezondheid
Excellent Vgood Good Fair Poor
0.11646107 0.33267012 0.39209444 0.13397002 0.02480435 In de laatste regel delen we de gehele (absolute) frequentietabel door het totaal aantal waarnemingen om zo de relatieve frequenties te krijgen.
De volgende code geeft het staafdiagram:
Een voorbeeld is een classificatie van de historische waarde van archeologische vondsten op een schaal van 1 tot 5.
Stel dat je de tijdsduur van een bepaald proces meet, maar je kunt om praktische redenen maar 1 keer per uur meten. Dan zal de tijdsduur—op zich een continue variabele—alleen discrete waarden aannemen (1 uur, 2 uur , … ). Hoe grover de resolutie van je meting, hoe meer je geneigd zou zijn om de gegevens als discreet op te vatten.
Of je bepaalde klassen als natuurlijk geordend beschouwt, hangt erg af van de focus van je onderzoek. Bedrijfsnamen hebben bijvoorbeeld een alfabetische ordening. Meestal is die volledig irrelevant en zou je die naam als nominale variabele beschouwen. Maar wat als je wilt onderzoeken of bedrijven die bovenaan de lijst staan in een bedrijvengids vaker als eerste door klanten worden gebeld?
Het volgende korte scriptje maakt eerst het (bimodale) histogram en plot daarna gewicht (Weight) tegen leeftijd AgeMonths.
Code:
library(NHANES) # library NHANES inladen
data(NHANES) # dataset in dataframe zettenCode:
Code:
Aan deze plot kun je aflezen dat vrijwel alle personen die minder dan 40 kg wegen jonger zijn dan 160 maanden. De bimodaliteit van de gegevens komt dus doordat de populatie grofweg uit twee groepen bestaat: volwassenen met een modus rond 75kg en kinderen met een modus rond 20kg. (Die onderverdeling is natuurlijk niet scherp: jongeren in de puberteit vallen tussen de wal en het schip.)
We kunnen dat verder demonstreren door aparte histogrammen te tekenen voor volwassenen en voor kinderen/jongeren. We trekken de grens bij 15 jaar.
library(NHANES) # library NHANES inladen
data(NHANES) # dataset in dataframe zetten
grens <- 15 # grens in jaren
volwassen <- (NHANES$AgeMonths > grens*12)
hist(NHANES$Weight[volwassen], breaks = 40, main = NULL) # histogram volwassenenhist(NHANES$Weight[!volwassen], breaks = 40, main = NULL) # histogram van de jeugdHet gemiddelde:
\[\bar{x} = \frac{\sum^n_{i=1}x_i}{n} = \frac{(3{,}5+2{,}4+3{,}4+3{,}9+2{,}7)\,\textrm{in}}{5} = 3{,}18 \, \textrm{in}.\]
De variantie:
\[ \begin{align} V_X & = \frac{\sum_i^n \left(x_i - \overline{x}\right)^2}{n-1}\\ &= \frac{\sum_i^n \left(3{,}5\,\textrm{in} - 3{,}18\,\textrm{in}\right)^2 + \left(2{,}4\,\textrm{in} - 3{,}18\,\textrm{in}\right)^2 + \ldots}{5 - 1}\\ &= 0{,}38\,\textrm{in}^2.\\ \end{align} \]
(Let op dat je tussendoor niet afrondt!)
De standaarddeviatie:
\[s_X = \sqrt{V_X} = \sqrt{0{,}377}\,\textrm{in} = 0{,}61\,\textrm{in}.\]
Verifiëren in R:
data <- c(3.5, 2.4, 3.4, 3.9, 2.7)
mean(data)[1] 3.18var(data)[1] 0.377sd(data)[1] 0.6140033Als we van iedere lengte 2 in aftrekken, verschuiven alle datapunten evenveel. Het gemiddelde neemt dus met 2 inches af en wordt 1,2 in.
Als alle punten evenveel opschuiven, blijft de spreiding van de gegevens gelijk. (Alle afstanden tussen punten blijven onveranderd, en het histogram blijft even breed.) Daarom moeten ook variantie en standaarddeviatie onveranderd blijven.
Verifiëren in R:
data_verschoven <- data - 2
mean(data_verschoven)[1] 1.18var(data_verschoven)[1] 0.377sd(data_verschoven)[1] 0.6140033Als alle waarden in cm worden uitgedrukt, worden ze allemaal een factor 2,54 groter. Dat betekent dat ook het gemiddelde een factor 2,54 groter wordt. Je ziet dit vanzelf als je invult dat \(1\,\text{in} = 2{,}54\,\text{cm}\):
\[\overline{x} = 3{,}18\,\text{in} = 3{,}18 \times (2{,}54\,\text{cm}) = 8{,}1\,\text{cm}.\]
Hetzelfde geldt voor de standaarddeviatie: als alle getallen een factor 2,54 groter worden, dan worden ook alle afstanden diezelfde factor groter, en de breedte van de verdeling ook. Ook dat volgt direct als we invullen dat \(1\,\text{in} = 2{,}54\,\text{cm}\):
\[s_X = 0{,}614\,\text{in} = 0{,}614\times(2{,}54\,\text{cm}) = 1{,}56\,\text{cm}.\]
De variantie is iets ingewikkelder. De variantie heeft dimensie lengte-in-het-kwadraat, en eenheid \(\text{in}^2\). Als alle waarden een factor 2{,}54 groter worden, dan wordt de variantie dus een factor \(2{,}54^2\) groter! Je kunt dat ook zien in de formule voor \(V_X\): de residuen worden gekwadrateerd, en dus ook de factor \(2{,}54\).
Maar je krijgt het juiste resultaat ook door weer gewoon \(1\,\text{in} = 2{,}54\,\text{cm}\) in te vullen:
\[V_X = 0{,}377\,\text{in}^2 = 0{,}377\times(2{,}54\,\text{cm})^2 = 2{,}43\,\text{cm}^2.\]
Verifiëren in R:
data_cm <- data*2.54
mean(data_cm)[1] 8.0772var(data_cm)[1] 2.432253sd(data_cm)[1] 1.559568Populatie: Sterren van het Virgo Cluster. Eenheden: Individuele sterren uit het cluster. Variabele: Temperatuur van een ster. Populatieparameter: De standaarddeviatie van de verdeling van temperaturen van sterren in de populatie.
Populatie: Bierflesjes geproduceerd door de fabriek. Eenheden: Individuele flesjes uit de fabriek. Variabele: Graad van vervuiling. Populatieparameter: Gemiddelde vervuilingsgraad in de “populatie” van geproduceerde flesjes.
Populatie: Kinesine-1 in neurons. Eenheden: Individuele motoren van Kinesine-1. Variabele: Stapsnelheid. Populatieparameter: Gemiddelde stapsnelheid van de populatie kinesines.
Populatie: Nederlandse bevolking. Eenheden: Individuele Nederlanders. Variabele: Steun voor het kabinetsbeleid (bijv. “Ja”/“Nee”). Populatieparameter: Proportie van de populatie dat het kabinetsbeleid steunt.
Toevallige fout. Ieder cijfer hangt af van de studentassistent die de opdracht toevallig nakijkt.
Systematische fout. Door de manier waarop de steekproef werd genomen (via de lijst van Literary Digest) was deze sterk gebiast. Dat levert een systematische fout op.
Systematische fout. De tijden wijken namelijk systematisch 4 min af van de werkelijke waarde.
Toevallige fout. Er is variatie in de concentraties door toevallige afwijkingen bij het pipetteren.
Waarnemersbias ligt het meest voor de hand, omdat de psychiater de toestand van zijn eigen cliënten beoordeelt en daar mogelijk een verwachting of vooroordeel over heeft. Overlevingsbias kan een rol spelen als bij sommige cliënten de behandeling tussentijds wordt afgebroken; in dat geval zullen we alleen de klachten beoordelen van een specifieke selectie van de cliënten.
Vrijwilligersbias speelt daar een enorme rol, want de mensen die de moeite nemen om te stemmen zijn geen willekeurige steekproef uit de populatie. Stand.nl weet dit heel goed: deze disclaimer staat op hun website bij de uitslag:
Overlevingsbias. Bedrijven die tijdens de crisis failliet zijn gegaan staan mogelijk in 2024 niet meer ingeschreven bij de Kamer van Koophandel. Het effect van de steun op die bedrijven wordt in het onderzoek dus niet meegenomen. Dat levert bias op.
Non-respons-bias: typisch vult maar ongeveer 20% van de studenten de enquête in. Niemand weet welk effect dat heeft op de gemiddelde cijfers. (Vul jij de enquête na deze cursus wel in? Een grotere steekproef vermindert de bias.)
Overlevingsbias. Deze CEO schat het effect van stoppen met je studie in op basis van een heel bijzondere selectie personen: succesvolle managers in de ICT. Dat levert een vertekend beeld op. Het perspectief van de drop-outs die géén CEO zijn geworden wordt niet meegenomen.
Mogelijk stopten deze succesvolle ICT-ers met hun studie omdat ze concrete mogelijkheden zagen op de arbeidsmarkt, of hadden ze een bijzonder talent. Dat maakt het niet per se een goed idee voor anderen om hun voorbeeld te volgen.
Het volgende scriptje werkt hiervoor:
# parameters
aantal_plotjes <- 20
aantal_selecteren <- 10
# vector met nummers plotjes
nummers_plotjes <- seq(1, aantal_plotjes)
# selecteer plotjes die bemest gaan worden
(bemesten <- sample( nummers_plotjes, aantal_selecteren) )Zie het volgende script:
library(NHANES)
data(NHANES)
# parameters
n_steekproef <- 30 # grootte van de steekproef
# steekproef
steekproef <- sample(
NHANES$SleepHrsNight, n_steekproef
)
gemiddelde <- mean(steekproef, na.rm = TRUE)
print(steekproef)
print(gemiddelde)Merk op dat er op veel plekken NA is ingevuld — dat zijn missende gegevens.
–
De verschillende schattingen zijn verschillend door steekproevenvariabiliteit: toevallige verschillen in de samenstelling van steekproeven uit dezelfde populatie.
Omdat we een aselecte steekproef hebben genomen, hebben de schattingen geen bias; de schattingen zijn nauwkeurig / zuiver. Dat wil zeggen dat de schattingen gemiddeld juist zijn. We kunnen het populatiegemiddelde dus schatten door het gemiddelde te bepalen van de schattingen in het histogram. Op het oog is dat ongeveer 6,9 uur.
We kunnen dat gemiddelde van die schattingen ook rechtstreeks aan R vragen door deze regel toe te voegen aan de code:
mean(gemiddelden)Het gemiddelde van de hele populatie krijgen we als volgt:
mean(slaapuren)Als het goed is, liggen deze getallen dicht bij elkaar, rond 6,9 uur.
De onzekerheid of toevallige fout in één schatting hangt samen met de spreiding van de herhaalde schattingen. Een goede optie is dus om de standaarddeviatie van de schattingen te gebruiken:
sd(gemiddelden)Het precieze resultaat hangt af van de toevallige steekproeven die jij hebt getrokken, maar zou dichtbij 0,25 moeten liggen. Dat wil zeggen: typisch ligt één schatting blijkbaar zo’n 0,25 van de werkelijke waarde van de populatieparameter af.
Kansexperiment: Het kruisen van twee heterozygote dieren en het bepalen van het genotype van een nakomeling.
Kansvariabele: Het genotype van de nakomeling.
Kansruimte: De verzameling {aa, AA, aA}.
Gebeurtenis: \(X\) = aa.
Kansexperiment: Het tellen van het aantal teken op een ree aangetroffen in het bosgebied.
Kansvariabele: Aantal teken.
Kansruimte: Alle niet-negatieve gehele getallen.
Gebeurtenis: \(X > 0\).
Kansexperiment: Het vaccineren van een persoon en kijken of TTS optreedt.
Kansvariabele: Een Boolean variabele die aangeeft of TTS optrad.
Kansruimte: {TRUE, FALSE}
Gebeurtenis: \(X\) = TRUE.
Kansexperiment: Het vangen, markeren en teruggooien van 2000 brasems; opnieuw vangen van 2000 brasems; en het tellen van het aantal gemarkeerde brasems.
Kansvariabele: Aantal teruggevangen gemarkeerde brasems.
Kansruimte: Gehele getallen van 0 tot 2000.
Gebeurtenis: \(X = 0\).
Gebruik voor de absolute frequenties:
table(resultaten)resultaten
1 2 3 4 5 6
166474 166755 166992 166813 166001 166965 Voor de relatieve frequenties:
table(resultaten)/aantal_keer_gooienresultaten
1 2 3 4 5 6
0.166474 0.166755 0.166992 0.166813 0.166001 0.166965 Jouw uitkomsten zullen hier door steekproevenvariabiliteit iets van afwijken.
Nee, want dan moest de relatieve frequentie gelijk zijn aan \(1/6 = 0{,}16666\ldots\).
Die kans is 0, want een miljoen is niet deelbaar door 6. Dat is één reden waarom de relatieve frequentie zelfs bij een enorm grote steekproef zelden exact \(\frac{1}{6}\) zal zijn: de meeste getallen zijn niet deelbaar door 6. Maar zelfs als aantal_keer_gooien 6 miljoen is en dus wél deelbaar door 6, is de kans heel klein dat we precies 1 miljoen keer een 6 vinden, en niet een paar meer of minder.
–
Venn-diagram:
Ja, want 5 is oneven én groter dan 4.
In dit geval geldt: \(\textrm{Pr}\!\left[A \text{ en } B\right] = \textrm{Pr}\!\left[X = 5\right] = \frac{1}{6}\).
Exclusief, want er wordt niet bedoeld dat één en dezelfde cursus zowel een studielast van 7,5 ECTS als een veelvoud daarvan kan hebben.
Inclusief, want je mag vast ook feedback geven over zaken die anders én beter zouden kunnen.
Inclusief, want het is vast ook niet toegestaan dat iemand met een managementfunctie lid én voorzitter wordt.
Exclusief, want je krijgt maar één beoordeling, die niet zowel numeriek als categorisch (V / ONV) kan zijn.
Venn-diagram:
{1, 3, 5, 6}
Omdat het een eerlijke dobbelsteen is en er vier uitkomsten onder de gebeurtenis vallen: \(\textrm{Pr}\!\left[A \text{ of } B\right] = \frac{4}{6}\).
\(\textrm{Pr}\!\left[A\right] = \frac{3}{6} = \frac{1}{2}.\)
\(\textrm{Pr}\!\left[B\right] = \frac{2}{6} = \frac{1}{3}.\)
Zie Oefening 22.3: \(\textrm{Pr}\!\left[A \text{ en } B\right] = \frac{1}{6}.\)
\(\textrm{Pr}\!\left[A|B\right] = \dfrac{\textrm{Pr}\!\left[A \text{ en } B\right]}{\textrm{Pr}\!\left[B\right]} = \dfrac{\frac{1}{6}}{\frac{1}{3}} = \frac{1}{2}.\)
\(\textrm{Pr}\!\left[B|A\right] = \dfrac{\textrm{Pr}\!\left[A \text{ en } B\right]}{\textrm{Pr}\!\left[A\right]} = \dfrac{\frac{1}{6}}{\frac{1}{2}} = \frac{1}{3}.\)
\(\textrm{Pr}\!\left[B|A\right]\) is de kans op \(A\) en \(B\) relatief ten opzichte van de kans op \(A\), \(\textrm{Pr}\!\left[A|B\right]\) de kans op \(A\) en \(B\) relatief ten opzichte van de kans op \(B\). Zolang de kans op \(A\) niet gelijk is aan de kans op \(B\) zijn beide conditionele kansen dus verschillend.
Het is handig om R als rekenmachine gebruiken. We voeren eerst netjes alle getallen in:
# Definieer de populatiegrootte en aantallen
nederlanders <- 17.6*10^6
diabetes <- 1.08*10^5
hartfalen <- 2.5*10^5
beide <- 4*10^3
# Bereken de aantallen voor ieder compartiment
alleen_diabetes <- diabetes - beide
alleen_hartfalen <- hartfalen - beide
geen_van_beide <- nederlanders - (alleen_diabetes + alleen_hartfalen + beide)Het Venn-diagram ziet er dan als volgt uit:
De kans dat een willekeurige Nederlander hartfalen heeft is:
(prob_H <- hartfalen / nederlanders)[1] 0.01420455De kans dat een willekeurige Nederlander diabetes type 1 heeft is:
(prob_D <- diabetes / nederlanders)[1] 0.006136364De kans dat een diabetespatiënt ook hartfalen heeft:
(prob_H_gegeven_D <- beide / diabetes)[1] 0.03703704Dit is precies wat bedoeld wordt met de kans \(\textrm{Pr}\!\left[H | D\right]\).
Om te bepalen of het risico op hartfalen groter is voor mensen met diabetes, berekenen we de verhouding tussen \(\textrm{Pr}\!\left[H | D\right]\) en \(\textrm{Pr}\!\left[H\right]\):
(risico_verhoging <- prob_H_gegeven_D / prob_H)[1] 2.607407Mensen met diabetes hebben volgens deze berekening een 2,6 keer zo hoog risico op hartfalen vergeleken met de algemene bevolking.
In Oefening 22.6 was \(\textrm{Pr}\!\left[A\right] = \textrm{Pr}\!\left[A | B\right] = \frac{1}{2}\). Deze gebeurtenissen zijn dus onafhankelijk! Dit blijkt ook uit het feit dat \(\textrm{Pr}\!\left[B\right] = \textrm{Pr}\!\left[B | A\right] = \frac{1}{3}\).
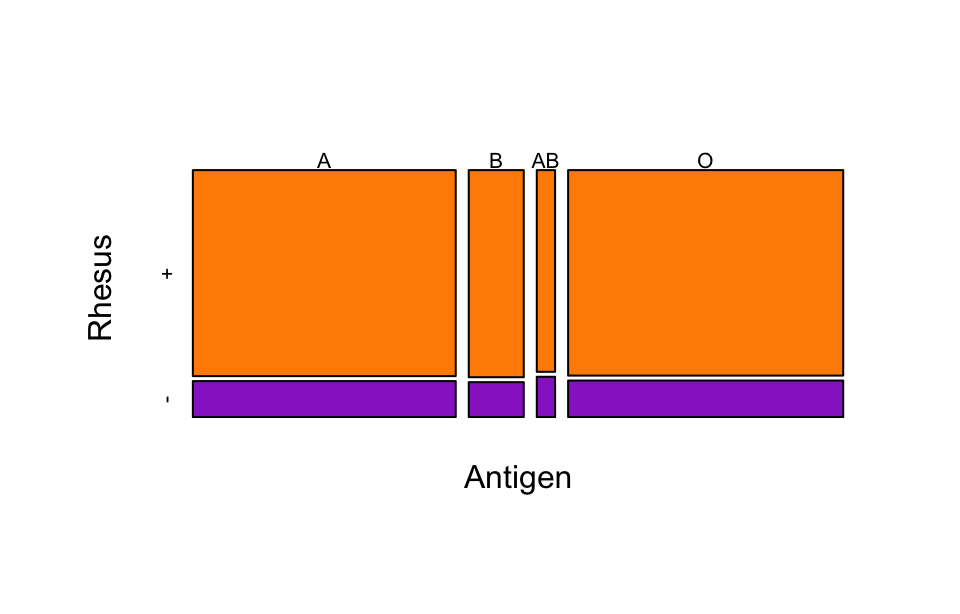
Aflezen van de figuur: \(\textrm{Pr}\!\left[\text{O+}\right] = 0{,}382\).
\(\textrm{Pr}\!\left[\text{O}\right] = \textrm{Pr}\!\left[\text{O+}\right] + \textrm{Pr}\!\left[\text{O-}\right] = 0{,}382 + 0{,}068 = 0{,}45\).
We tellen de kansen van alle Rh+ bloedgroepen op:
\[ \begin{align} \textrm{Pr}\!\left[\text{+}\right] &= \textrm{Pr}\!\left[\text{O+}\right] + \textrm{Pr}\!\left[\text{A+}\right] + \textrm{Pr}\!\left[\text{B+}\right] + \textrm{Pr}\!\left[\text{AB+}\right]\\ &= 0{,}382 + 0{,}366 + 0{,}077 + 0{,}025 = 0{,}85. \end{align} \]
Als antigen en rhesusfactor onafhankelijk zouden zijn, dan zou de vermenigvuldigingsregel gelden:
\[ \begin{align} \textrm{Pr}\!\left[\text{O+}\right] &= \textrm{Pr}\!\left[\text{O en +}\right] = \textrm{Pr}\!\left[\text{O}\right]\cdot\textrm{Pr}\!\left[\text{+}\right]\\ &= 0{,}45 \times 0{,}85\\ &= 0{,}383. \end{align} \]
Daarmee komen we heel dicht bij de werkelijke waarde van 0,382! Blijkbaar zijn gebeurtenissen O en + vrijwel onafhankelijk. Bij berekeningen aan bloedgroepen maken we slechts een kleine fout als we een kansmodel gebruiken dat aanneemt dat antigeen en rhesusfactor onafhankelijk zijn.
De ABO-bloedgroep en de rhesusfactor worden bepaald door genen die op verschillende chromosomen liggen. Die segregeren dus onafhankelijk van elkaar en linkage is dus niet te verwachten. Kleine associaties kunnen wel optreden, bijvoorbeeld als bepaalde etnische bevolkingsgroepen onderling verschillen in hun proporties.
Het plaatje ziet er mooi uit, maar geeft de getallen niet helder weer. De oppervlakte van de bloeddruppels is niet in proportie met het percentage dat erbij hoort. Dat is misleidend: het lijkt er bijvoorbeeld niet op dat de kans op O+ maar liefst 76 keer zo groot is als de kans op AB-.
Deze figuur geeft de verhoudingen beter weer:
Maar een mosaicplot ligt meer voor de hand:
::: {.cell}
::: {.cell-output-display}
{width=100%}
:::
:::Nee, dat klopt niet. Je mag de kansen hier niet optellen. Dat zou betekenen dat de kans op een prijs na 10 keer meedoen gelijk zou zijn aan \(10\times \frac{1}{10} = 1\), alsof je na 10 keer zeker zou zijn van een prijs.
Het probleem is dat de twee gebeurtenissen (eerste keer prijs en tweede keer prijs) elkaar niet uitsluiten. De optelregel is dan niet van toepassing.
Ja, dat klopt. De gebeurtenissen O+, A+, B+ en AB+ sluiten elkaar uit en dus geldt de optelregel.
Als \(A\) en \(B\) elkaar uitsluiten is \(\textrm{Pr}\!\left[A \textrm{ en } B\right]\) gelijk aan 0.
Je kunt in dat geval ook de algemene regel gebruiken, want als je in de algemene regel \(\textrm{Pr}\!\left[A \textrm{ en } B\right] = 0\) invult krijg je de optelregel. Voor gebeurtenissen die elkaar uitsluiten komen beide regels dus op hetzelfde neer.
Een bacterie is resistent als deze mutatie 1 heeft (gebeurtenis \(M_1\)) of mutatie 2 (gebeurtenis \(M_2\)).
Gegeven is: \[ \begin{gather} \textrm{Pr}\!\left[M_1\right] = 0{,}3, \\ \textrm{Pr}\!\left[M_2\right] = 0{,}4, \\ \textrm{Pr}\!\left[M_1 \text{ en } M_2\right] = 0{,}15. \end{gather} \]
Regel Vergelijking 22.9 levert dan op:
\[\textrm{Pr}\!\left[M_1 \text{ of } M_2\right] = \textrm{Pr}\!\left[M_1\right] + \textrm{Pr}\!\left[M_2\right] - \textrm{Pr}\!\left[M_1 \text{ en } M_2\right] = 0{,}3 + 0{,}4 - 0{,}15 = 0{,}55.\]
Venn-diagram:
Als je \(\textrm{Pr}\!\left[A\right]\), \(\textrm{Pr}\!\left[B\right]\) en \(\textrm{Pr}\!\left[C\right]\) optelt, worden de overlappende delen tussen \(A\) en \(B\), tussen \(B\) en \(C\), en tussen \(A\) en \(C\) dubbelgeteld. Die trek je er dus vanaf. Maar, door dat te doen trek je het gedeelte \(A\) en \(B\) en \(C\) (de overlap tussen de drie cirkels) er drie keer vanaf. Om te zorgen dat dat deel ook één keer wordt meegeteld, moet die er weer bij opgeteld worden. Het resultaat is:
\[ \begin{align} \textrm{Pr}\!\left[A \text{ of } B \text{ of } C\right] = & \ \textrm{Pr}\!\left[A\right] + \textrm{Pr}\!\left[B\right] + \textrm{Pr}\!\left[C\right] \\ & - \textrm{Pr}\!\left[A \text{ en } B\right] - \textrm{Pr}\!\left[B \text{ en } C\right] - \textrm{Pr}\!\left[A \text{ en } C\right]\\ & + \textrm{Pr}\!\left[A \text{ en } B \text{ en } C\right]. \end{align} \]
De eerdere resultaten waren (afgerond): \[ \begin{gather} \textrm{Pr}\!\left[H | D\right] = 0{,}037,\\ \textrm{Pr}\!\left[H\right] = 0{,}014,\\ \textrm{Pr}\!\left[D\right] = 0{,}0061. \\ \end{gather} \] De regel van Bayes zegt: \[\textrm{Pr}\!\left[D | H\right] = \textrm{Pr}\!\left[H | D\right] \textrm{Pr}\!\left[D\right]/\textrm{Pr}\!\left[H\right].\] Invullen:
(prob_D_gegeven_H <- prob_H_gegeven_D * prob_D / prob_H)[1] 0.016Het model nam aan dat iedere nucleotide onafhankelijk met kans \(1 - p\) een C,T, of A was. Dat geldt ook voor het nucleotide direct na de G. De kans op lengte 1 is dus \(\textrm{Pr}\!\left[X = 1\right] = 1 - p\).
De kans dat de volgende letter een G is, is volgens het model \(p\). De kans dat de letter dáárna geen G is, is \(1 - p\). Omdat beide gebeurtenissen onafhankelijk zijn (volgens het model) mogen we ze vermenigvuldigen: \(\textrm{Pr}\!\left[X = 2\right] = p (1 - p)\).
De kans dat de volgende letter een G is, is volgens het model \(p\). De kans dat de letter daarna wéér een G is, is \(p\). De kans dat de letter daarna geen G is, is \(1 - p\). Omdat alle gebeurtenissen onafhankelijk zijn (volgens het model) mogen we ze vermenigvuldigen: \(\textrm{Pr}\!\left[X = 3\right] = p \cdot p \cdot (1 - p) = p^2 (1 - p)\).
Invullen:
\(\textrm{Pr}\!\left[X = 1\right] = 1 - p = 0{,}795\),
\(\textrm{Pr}\!\left[X = 2\right] = p (1 - p) = 0{,}163\),
\(\textrm{Pr}\!\left[X = 3\right] = p^2 (1 - p) = 0{,}0334\),
\(\textrm{Pr}\!\left[X = 4\right] = p^3 (1 - p) = 0{,}00685\), etc.
De paarse lijn komt aardig in de buurt van de echte gegevens. G-tracts van de lengte die we in chromosoom 11 vinden zouden we dus ook ongeveer verwachten op basis van toeval. Er is dus geen specifieke biologische functie of sterke natuurlijke selectie nodig om de gevonden G-tracts te verklaren.
Als je in detail kijkt, is de kans op een lengte van 1 iets kleiner dan verwacht, en de kans op langere G-tracts juist iets groter dan verwacht. Dat duidt erop dat de neiging van G’s om gevolgd te worden door een andere G net iets groter is dan je zou verwachten als de opeenvolgende nucleotiden volledig onafhankelijk waren. Dat zou je verder kunnen onderzoeken; je zou je kunnen uitzoeken of dat effect komt door coderende sequenties, of door niet-coderende sequenties (of beiden).
\(\textrm{Pr}\!\left[K\right]\) is de prevalentie. Gegeven is dat \(\textrm{Pr}\!\left[K\right] = 0{,}001\). Dat zijn naar verwachting dus \(120\,000\times\textrm{Pr}\!\left[K\right] = 120\) vrouwen die borstkanker hebben.
\(\textrm{Pr}\!\left[G\right] = 1 - \textrm{Pr}\!\left[K\right] = 0{,}999\). Dat komt neer op \(120\,000\times \textrm{Pr}\!\left[G\right] = 119\,880\) gezonde vrouwen.
\(\textrm{Pr}\!\left[P | K\right]\) is de sensitiviteit. Gegeven is dat \(\textrm{Pr}\!\left[P | K\right] = 0{,}9\).
Als we alle vrouwen van 30 jaar screenen, verwachten we dus \(120\times \textrm{Pr}\!\left[P | K\right] = 108\) keer borstkanker te ontdekken.
\(\textrm{Pr}\!\left[N | G\right]\) is de specificiteit. Die is ook gegeven: \(\textrm{Pr}\!\left[N | G\right] = 0{,}95\).
\(\textrm{Pr}\!\left[P | G\right] = 1 - \textrm{Pr}\!\left[N | G\right] = 0,05\). We testen naar verwachting 119880 gezonde vrouwen. (Zie onderdeel b.) De test valt dus onterecht positief uit bij \(119\,880 \times \textrm{Pr}\!\left[ P | G \right] = 5994\) vrouwen. Dat zijn er veel meer dan de 108 terecht positieve testen!
\(\textrm{Pr}\!\left[P\right] = 0{,}05085\).
Naar verwachting krijgen dus \(120\,000\times\textrm{Pr}\!\left[P\right] = 6102\) vrouwen een positief testresultaat.
Gebruik de regel van Bayes:
\[\textrm{Pr}\!\left[K | P\right] = \textrm{Pr}\!\left[P | K\right] \textrm{Pr}\!\left[K\right]/\textrm{Pr}\!\left[P\right].\]
De kansen aan de rechterkant heb je uitgerekend in onderdelen a, c, en f, dus je kunt de formule invullen.
Het resultaat is
\[\textrm{Pr}\!\left[K | P\right] = \textrm{Pr}\!\left[P | K\right] \textrm{Pr}\!\left[K\right]/\textrm{Pr}\!\left[P\right] = 0{,}9\times 0{,}001 / 0{,}05085 = 0{,}0177.\]
Dat betekent: als de test positief terugkomt, is de kans dat je daadwerkelijk borstkanker hebt nog steeds maar \(0{,}018\), minder dan 2%!
Een groot nadeel van vroegtijdig screenen is het grote aantal fout-positieve testresultaten. De overgrote meerderheid van de vrouwen die naar het ziekenhuis worden doorverwezen hebben in werkelijkheid geen borstkanker, wat leidt tot veel onnodige paniek en veel overbodige medische procedures.
De screening-paradox treedt op wanneer een test een hoge sensitiviteit en specificiteit heeft, maar de prevalentie van de ziekte laag is. Hoewel de test goed werkt, is het absolute aantal fout-positieven dan vaak veel groter dan het aantal correct-positieven, waardoor de kans dat een positief geteste persoon daadwerkelijk ziek is verrassend laag blijft.
Het volgende script in R voert alle berekeningen uit:
# Gegeven waarden
prevalentie <- 0.001 # P(K)
sensitiviteit <- 0.9 # P(P | K)
specificiteit <- 0.95 # P(N | G)
totaal_vrouwen <- 120000
# Berekeningen
prob_k <- prevalentie
prob_g <- 1 - prevalentie
prob_n_gegeven_g <- specificiteit
prob_p_gegeven_g <- 1 - prob_n_gegeven_g
prob_p_gegeven_k <- sensitiviteit
prob_p <- prob_p_gegeven_g * prob_g + prob_p_gegeven_k * prob_k
aantal_k <- totaal_vrouwen * prob_k
aantal_g <- totaal_vrouwen * prob_g
aantal_true_p <- aantal_k * prob_p_gegeven_k
aantal_false_p <- aantal_g * prob_p_gegeven_g
aantal_p <- aantal_true_p + aantal_false_p
# kans op borstkanker gegeven positieve test:
(prob_k_gegeven_p <- prob_p_gegeven_k * prob_k / prob_p) [1] 0.01769912Reken eerst de kansen uit. De uitkomst van de eerste munt noemen we \(X_1\) en die van de tweede munt \(X_2\). De uitkomsten zijn K (voor kop) of M (voor munt).
De kans op 0 keer kop: \[\textrm{Pr}\!\left[0\right] = \textrm{Pr}\!\left[X_1 = \text{ M en } X_2 = \text{M}\right] = \left(\frac{1}{2}\right)^2 = \frac{1}{4}.\] De kans op 1 keer kop \[ \begin{align} \textrm{Pr}\!\left[1\right] &= \textrm{Pr}\!\left[\left(X_1 = \text{ M en } X_2 = \text{K}\right)\text{ of }\left(X_1 = \text{ K en } X_2 = \text{M}\right)\right]\\ & = 2 \times \left(\frac{1}{2}\right)^2\\ & = \frac{1}{2}. \end{align} \] De kans op 2 keer kop: \[\textrm{Pr}\!\left[2\right] = \textrm{Pr}\!\left[X_1 = \text{ K en } X_2 = \text{K}\right] = \left(\frac{1}{2}\right)^2 = \frac{1}{4}.\]
Dat levert de volgende plot op:
\(\textrm{Pr}\!\left[\texttt{aa}\right] = \textrm{Pr}\!\left[X_1 = \texttt{a}\text{ en } X_2 = \texttt{a}\right] = \left(\frac{1}{2}\right)^2 = \frac{1}{4}.\)
\(\textrm{Pr}\!\left[\texttt{aA}\right] = \textrm{Pr}\!\left[\left(X_1 = \texttt{a}\text{ en } X_2 = \texttt{A}\right)\text{ of }\left(X_1 = \texttt{A}\text{ en } X_2 = \texttt{a}\right)\right] = 2\times \left(\frac{1}{2}\right)^2 = \frac{1}{2}.\)
\(\textrm{Pr}\!\left[\texttt{AA}\right] = \textrm{Pr}\!\left[X_1 = \texttt{A}\text{ en } X_2 = \texttt{A}\right] = \left(\frac{1}{2}\right)^2 = \frac{1}{4}.\)
Het is precies dezelfde berekening!
Waar het in Oefening 23.1 ging over het aantal keer “kop” gaat het hier om het aantal keer A, maar verder zijn de modellen gelijk.
We berekenen de cumulatieve kansen:
\(F_X(0) = \textrm{Pr}\!\left[0\right] = \frac{1}{4}.\)
\(F_X(1) = \textrm{Pr}\!\left[0\right] + \textrm{Pr}\!\left[1\right] = \frac{3}{4}.\)
\(F_X(2) = \textrm{Pr}\!\left[0\right] + \textrm{Pr}\!\left[1\right] + \textrm{Pr}\!\left[2\right] = 1.\)
De vakjes zijn 1 eenheid breed en 0,05 hoog. De oppervlakte is dus \(1\times 0{,}05 = 0{,}05\). Daarom staat één vakje dus voor een kans van 0,05.
Paneel (a): De kans is ongeveer 0,5. Dit is duidelijk te zien, omdat het ingekleurde gebied begint bij de piek van de verdeling en de verdeling er symmetrisch uitziet.
Paneel (b):
De kans is ongeveer 0,25. Dit kun je bepalen door het aantal hele hokjes in het paarse gebied te schatten (ongeveer 5), en het totaal te vermenigvuldigen met 0,05.
Paneel (c):
De paarse oppervlakjes van (b) en (c) zijn samen precies even groot als die van (a). De oppervlakte van (c) is dus 0,5 - 0,25 = 0,25. Natuurlijk kun je ook weer gewoon hokjes tellen.
Paneel (d):
De kans is ongeveer 0,5. We hebben hier twee keer de oppervlakte van het paarse gebied in (c).
Gebruik de complementregel:
\(\textrm{Pr}\!\left[X < 170 \text{ of } X > 182\right] = 1 - \textrm{Pr}\!\left[170 < X < 182\right] = 1 - 0{,}59 = 0{,}41\).
De kans van het deel \(X < 170\) lijkt ongeveer even groot als die van \(X > 182\). De kans \(\textrm{Pr}\!\left[X < 170\right]\) is dus ongeveer de helft van 0,41. Inderdaad: een exacte meting van het oppervlak (die jij niet kunt nadoen) geeft 0,21.
–
Formule Vergelijking 20.7 uit Oefening 20.10 drukt uit hoe je het gemiddelde van een reeks getallen kunt uitrekenen op basis van de relatieve frequenties \(f(x)\) van alle uitkomsten \(x\).
In paragraaf Kansen als frequenties van een lange serie identieke kansexperimenten hebben we kansen \(\textrm{Pr}\!\left[X = x\right]\) gedefinieerd als relatieve frequentie van \(x\) in de uitkomsten van een (oneindig) lange reeks herhalingen van hetzelfde kansexperiment. Het gemiddelde van zo’n oneindig lange reeks kan dus met Vergelijking 20.7 worden berekend, waarbij we de kans \(\textrm{Pr}\!\left[X = x\right]\) invullen op de plek van \(f(x)\). Dat levert precies Vergelijking 23.2 op.
We moeten steeds iedere uitkomst vermenigvuldigen met zijn kans, en de resultaten optellen. Deze code voert de berekeningen uit:
aantal_gelegenheden <- 9
uitkomsten <- seq(0, aantal_gelegenheden) #alle mogelijke uitkomsten
kansen <- c( # de kansen die in de tabel van de opgave gegeven zijn
0.009, 0.054, 0.151, 0.244, 0.255,
0.177, 0.082, 0.024, 0.004,0.000
)
( E <- sum( kansen * uitkomsten) ) # verwachtingswaarde[1] 3.685We vervolgen het script van Oefening 23.7.
De afwijkingen van het gemiddelde
( afwijkingen <- uitkomsten - E ) [1] -3.685 -2.685 -1.685 -0.685 0.315 1.315 2.315 3.315 4.315 5.315De verwachtingswaarde E wordt zo van iedere uitkomst afgetrokken. Het resultaat is dus weer een vector.
Kwadrateren:
( afwijkingen_kwadraat <- afwijkingen^2 ) [1] 13.579225 7.209225 2.839225 0.469225 0.099225 1.729225 5.359225
[8] 10.989225 18.619225 28.249225Product:
( prod <- kansen * afwijkingen_kwadraat ) [1] 0.12221303 0.38929815 0.42872298 0.11449090 0.02530237 0.30607282
[7] 0.43945645 0.26374140 0.07447690 0.00000000Variantie:
( variantie <- sum(prod) )[1] 2.163775We hadden alle stappen ook in één regel kunnen uitvoeren:
( variantie <- sum( kansen * (uitkomsten - E)^2 ) )[1] 2.163775Standaarddeviatie:
( standaarddeviatie <- sqrt(variantie) )[1] 1.470978::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::
Omdat het gearceerde deel van de curve precies tot het gemiddelde loopt
en de normale verdeling symmetrisch is, moet de kans 0,5 zijn.::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::
De eerste vuistregel zegt dat de kans binnen 1 standaarddeviatie 0,68... is.
Buiten die grenzen ligt dus, afgerond, 1 - 0,68 = 0,32.
Maar daarbij worden beide staarten meegerekend, terwijl hier alleen de rechterstaart
gearceerd is. De oppervlakte is dus 0,32/2 = 0,16.::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::
De tweede vuistregel zegt dat de kans binnen 2 standaarddeviaties 0,954... is.
Buiten die grenzen ligt dus, afgerond, 1 - 0,954 = 0,046.
Maar daarbij worden beide staarten meegerekend, terwijl hier alleen de linkerstaart
gearceerd is. De oppervlakte is dus 0,046/2 = 0,023.::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::
De tweede vuistregel zegt dat de kans binnen 2 standaarddeviaties 0,954... is.
Buiten die grenzen ligt dus, afgerond, 1 - 0,954 = 0,046.
Dat is precies het gedeelte dat hier gearceerd is.Een hoofdomtrek van 38 cm is \(38\textrm{\,cm} - 34{,}5\textrm{\,cm} = 3{,}5\textrm{\,cm}\) boven het gemiddelde. Dat is \(3{,}5 / 1{,}75 = 2\) standaarddeviaties. Kortom, de berekening is \[ \text{aantal standaarddeviaties} = \frac{X - \mu_X}{\sigma_X} = \frac{38 - 34{,}5}{1{,}75} = 2.\]
Schets:
::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::
De tweede vuistregel zegt dat de kans binnen 2 standaarddeviaties 0,954... is.
Buiten die grenzen ligt dus, afgerond, 1 - 0,954 = 0,046.
Maar daarbij worden beide staarten meegerekend, terwijl hier alleen de rechterstaart
gearceerd is. De oppervlakte is dus 0,046/2 = 0,023.
Dus: de kans op een hoofdomtrek van meer dan 38cm is 0,023.::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::
De kans $\pr{Z < 0}$ is in het plaatje ingetekend. Die kans is 0,5.
Dus is $F_Z(0) = 0{,}5$.Uitvoeren code:
::: {.cell}
```{.r .cell-code}
pnorm(0)
```
::: {.cell-output .cell-output-stdout}
```
[1] 0.5
```
:::
:::::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::De kans $\pr{Z < -1}$ is in het plaatje ingetekend. Die kans is
volgens de vuistregels $(1 - 0{,}68)/2 = 0{,}16$.
Dus is $F_Z(-1) = 0{,}16$.Uitvoeren code:
::: {.cell}
```{.r .cell-code}
pnorm(-1)
```
::: {.cell-output .cell-output-stdout}
```
[1] 0.1586553
```
:::
:::::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::De kans $\pr{Z > 0{,}5}$ is in het plaatje ingetekend.
Met `pnorm()` kunnen we de kans in een linkerstaart berekenen,
maar nu willen we de kans in een rechterstaart. Er zijn twee oplossingen.
De eerste is om de complementregel te gebruiken:
$$ \pr{Z > 0{,}5} = 1 - \pr{Z < 0{,}5}.$$
De tweede optie is om van de *symmetrie* van de normale verdeling gebruik te maken:
$$\pr{Z > 0{,}5} = \pr{Z < -0{,}5}.$$
Beide opties zouden hetzelfde antwoord moeten geven.
Uitvoeren code:
::: {.cell}
```{.r .cell-code}
# optie 1: gebruik de complementregel:
1 - pnorm(0.5)
```
::: {.cell-output .cell-output-stdout}
```
[1] 0.3085375
```
:::
```{.r .cell-code}
# optie 2: gebruik de symmetrie:
pnorm(-0.5)
```
::: {.cell-output .cell-output-stdout}
```
[1] 0.3085375
```
:::
:::::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::De kans $\pr{-1 < Z < 1}$ is in het plaatje ingetekend.
Met `pnorm()` krijgen we de kans van de linkerstaart,
maar nu willen we de kans in een middenstuk.De oplossing is om eerst $\pr{Z < 1}$ uit te rekenen,
::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::
en daar het stuk $\pr{Z < -1}$ vanaf te trekken:
::: {.cell}
::: {.cell-output .cell-output-stdout}
```
NULL
```
:::
:::
Immers: $\pr{ -1 < Z < 1} = \pr{Z < 1} - \pr{Z < -1}.$
Uitvoeren code:
::: {.cell}
```{.r .cell-code}
pnorm(1) - pnorm(-1)
```
::: {.cell-output .cell-output-stdout}
```
[1] 0.6826895
```
:::
:::
De uitkomst had je kunnen voorspellen, want dit is precies
vuistregel 1.Schets:
De waarde 140 ligt ver in de staart, en dus is maar een heel klein deel van het oppervlak gearceerd. (Klik zo nodig even op het figuur om het te vergroten.) De kans moet dus erg klein zijn. Berekening:
1 - pnorm(140, mean = 100, sd = 15) # complementregel[1] 0.003830381De staarten van de \(t\)-verdeling zijn dikker. Extreme situaties waarbij een waarde veel groter is dan gemiddeld komen bij de \(t\)-verdeling dus vaker voor dan bij de normale verdeling.
Als het rekenmodel uitgaat van een normale verdeling schatten we de kans op extreme omstandigheden dus te laag in. Het model onderschat dan het risico op overstromingen.
De schets ziet er zo uit:
# Functie om een plot te maken van de kritieke waarde
crit_plot <- function(alpha) {
# Kritieke waarde van Z
Z_crit <- qnorm(1 - alpha / 2) # Bereken de kritieke waarde
# Coördinaten voor de pijlen
xstart <- (Z_crit + 3.5) / 2 # Beginpunt x-coördinaat
ystart <- dnorm(0) / 4 # Beginpunt y-coördinaat
xend <- Z_crit + (3.5 - Z_crit) / 7 # Eindpunt x-coördinaat
yend <- dnorm(xend) / 3 # Eindpunt y-coördinaat
proportie <- function(prop){
annotate(
"text",
x = 0, y = 0.15,
label = prop, # Dynamically include the variable
color = "black",
size = 5,
hjust = 0.5,
vjust = 0.5
)
}
# Plot de standaardnormale verdeling met staarten en annotaties
standaardNormaalGeneriek +
# Labels voor de assen
labs(
y = "Kansdichtheid",
x = expression(italic(Z))
) +
# Ingekleurd oppervlak onder de curve
oppervlak(-10, -Z_crit, data) +
oppervlak(Z_crit, 10, data) +
# Schaalinstellingen voor de x-as
scale_x_continuous(
breaks = c(-Z_crit, 0, Z_crit),
labels = c(
paste0(round(-Z_crit, 2), "..."),
"0",
paste0(round(Z_crit, 2), "...")
),
minor_breaks = NULL # Verwijder minor ticks
) +
# Kromme pijltjes toevoegen
geom_curve(
aes(x = -xstart, y = ystart, xend = -xend, yend = yend),
curvature = 0.2,
arrow = arrow(length = unit(0.2, "cm")),
color = "black",
size = 0.7
) +
geom_curve(
aes(x = xstart, y = ystart, xend = xend, yend = yend),
curvature = -0.2,
arrow = arrow(length = unit(0.2, "cm")),
color = "black",
size = 0.7
) +
# Annotaties toevoegen
annotate(
"text",
x = -xstart,
y = ystart + 0.02, # Plaats annotatie net boven de pijl
label = gsub("\\.", ",", round(alpha / 2, 3)),
color = "black",
hjust = 0.5,
size = 4
) +
annotate(
"text",
x = xstart,
y = ystart + 0.02, # Plaats annotatie net boven de pijl
label = gsub("\\.", ",", round(alpha / 2, 3)),
color = "black",
hjust = 0.5,
size = 4
) +
# Label in het midden
proportie(gsub("\\.", ",", round(1 - alpha, 2)))
}
# Voorbeeld: plot voor alpha = 0.1
crit_plot(0.1)NULLDe kritieke waarde (dus de grens van het gekleurde gebied) kun je berekenen als:
alfa <- 0.1
qnorm(1 - alfa/2)[1] 1.644854Als \(\alpha\) groter is, dan is het gearceerde deel van de plot groter. Dat wil zeggen dat de kritieke waarde kleiner moet zijn.
Als alfa gelijk is aan 1, dan is het volledige oppervlak onder de curve ingekleurd en moet de kritieke waarde dus 0 zijn.
alfa <- 1
qnorm(1 - alfa/2)[1] 0Code:
alfa <- 0.05
qt(1 - alfa/2, df = 2)[1] 4.302653Deze waarde is groter dan de kritieke waarde voor de normale verdeling (1,96). Dat is omdat de staarten van de \(t\)-verdeling dikker zijn.
Code:
alfa <- 0.05
qt(1 - alfa/2, df = 100)[1] 1.983972Deze waarde is kleiner dan voor df = 2. Bij grote aantal vrijheidsgraden gaat de \(t\)-verdeling steeds meer lijken op de standaardnormale verdeling. De kritieke waarde wordt dus kleiner als het aantal vrijheidsgraden groter wordt. De kritieke waarde is bij df = 100 al erg dicht bij die van de standaardnormale verdeling (1,96).
De puntschatter lijkt erop te wijzen dat de systolische bloeddruk door medicijn 1 omlaag gaat met 10 mmHg. Maar het 95%-BHI is breed en bevat zelfs positieve waarden. De schatting is dus erg onzeker en het bewijs zwak.
Nee. Het zou kunnen dat medicijn 1 geen effect heeft, maar we kunnen daar op basis van deze gegevens geen betrouwbare conclusies over trekken. Het BHI geeft aan dat we met 95% zekerheid enkel kunnen zeggen dat het ware effect tussen -25 en 5 mmHg in ligt.
Het 95%-BHI voor medicijn 2 loopt van -14 tot -6 mmHg. We zijn dus tenminste 95% zeker dat dit medicijn gemiddeld een negatief effect heeft op de bloeddruk. Dat is een sterke aanwijzing dat het medicijn effect heeft.
Dat kan. Het is altijd mogelijk dat de steekproef toevallig een extreem misleidend beeld geeft (als gevolg van steekproevenvariabiliteit). Maar, daarvoor is wel redelijk wat toeval nodig, want het komt maar één op de twintig keer voor (5%) dat de parameter niet in het 95%-BHI ligt, en het interval ligt ook nog eens op enige afstand van 0 mmHg af.
Dat meestal een 95%-BHI wordt berekend, en niet bijvoorbeeld een 50%-BHI, is deels een gewoonte. Er is niets magisch aan het getal 95.
Maar, een percentage in die buurt is geen slechte keuze.
Bij een 50%-BHI kan de parameter net zo goed binnen als buiten het interval liggen; we kunnen de waarden buiten het interval dus zeker niet uitsluiten. Andersom zou een 99,9%-BHI een enorm breed interval zijn. Zo’n breed interval zou ook niet zoveel zeggen; alleen dat je heel erg zeker bent dat de parameter ergens in dat enorme interval ligt. (“Ik ben 99,9% zeker dat de gemiddelde lengte van de Utrechtse student tussen 150cm en 230cm in ligt.” Duh!)
Het is voor de interpretatie nuttig als het BHI het interval is waarbinnen je de parameter redelijkerwijs mag verwachten. 95% is daarbij een aardig compromis.
Een schets van de verdeling; de zwarte pijl geeft de standaarddeviatie aan:
Het oppervlak is in de schets hierboven aangegeven. De grenzen liggen precies bij 2 standaarddeviaties van het gemiddelde. De vuistregel zegt dan dat de kans gelijk is aan \(1 - 0{,}954 = 0{,}046\).
De verwachtingswaarde van het steekproefgemiddelde is het populatiegemiddelde, dus \(110\textrm{\,cm}\).
De standaarddeviatie van het steekproefgemiddelde is
\(\sigma_{\overline{X}} = \cfrac{\sigma_{X}}{\sqrt{n}} = \cfrac{9\textrm{\,cm}}{\sqrt{9}} = 3\textrm{\,cm}.\)
Een schets van de steekproevenverdeling van het gemiddelde; de zwarte pijl geeft de standaarddeviatie \(\sigma_{\overline{X}}\) aan:
De situatie is in de schets hierboven ingetekend. De grenzen zijn weer precies twee standaarddeviaties boven en onder het gemiddelde, dus de kans is weer 0,046 (bijna 5%).
\(\overline{X} = 115{,}9\textrm{\,cm}\).
\(\sigma_{X} = 9\textrm{\,cm}\) (gegeven).
\(n = 9\).
\(\sigma_{\overline{X}} = \cfrac{\sigma_{X}}{\sqrt{n}} = 3\textrm{\,cm}\).
BHI: \(\overline{X} \pm 1{,}96 \sigma_{\overline{X}} = (115{,}9 \pm 5{,}9)\textrm{\,cm}\).
Kijk naar de formule:
\(\overline{X} \pm 1{,}96 \sigma_{\overline{X}} = \overline{X} \pm 1{,}96 \cfrac{\sigma_{X}}{\sqrt{n}}\).
Als \(\sigma_{\overline{X}}\) twee keer zo groot wordt, wordt het interval twee keer zo breed. Dat wil zeggen: als de spreiding in de populatie groter is, is de onzekerheid in het gemiddelde ook groter.
Kijk weer naar dezelfde formule. Als \(n\) twee keer zo groot is, wordt het interval \(\sqrt{2}\) keer zo smal. Grotere steekproeven leveren dus grotere precisie op, maar voor een 2 keer zo precieze schatting heb je een 4 keer zo grote steekproef nodig.
De onzekerheid in het gemiddelde hangt dus af van de spreiding in de populatie en de grootte van de steekproef.
De steekproefgrootte is \(n = 9\).
Het steekproefgemiddelde is \(\overline{X} = 115{,}9\textrm{\,cm}\).
De standaarddeviatie van de steekproef is \(s_X = 6{,}41\textrm{\,cm}\).
De standaardfout is \(\mathrm{SE}_{\overline{X}} = s_X / \sqrt{n} = 2{,}14\textrm{\,cm}\).
Het aantal vrijheidsgraden is \(\mathrm{df}= n - 1 = 8\).
De kritieke waarde \(t_{0{,}05 (2) \mathrm{8}}\):
alpha <- 0.05
qt(1 - alpha/2, df = 8)[1] 2.306004Ondergrens BHI: \(\overline{X} - t_{0{,}05 (2) \mathrm{8}} \mathrm{SE}_{\overline{X}} = 111{,}0\textrm{\,cm}\).
Bovengrens BHI: \(\overline{X} + t_{0{,}05 (2) \mathrm{8}} \mathrm{SE}_{\overline{X}} = 120{,}8\textrm{\,cm}\).
Het BHI is dus \([111{,}0\textrm{\,cm}; 120{,}8\textrm{\,cm}]\), oftewel \((115{,}9 \pm 4{,}9)\textrm{\,cm}\).
In een script:
# gegeven:
spanwijdte <- c(117, 106, 123, 111, 124, 120, 108 ,115, 119)
# berekening
n <- length(spanwijdte) # aantal datapunten
m <- mean(spanwijdte) # gemiddelde van steekproef
s_X <- sd(spanwijdte) # standaarddeviatie steekproef
SEM <- s_X/sqrt(n) # standaardfout van het gemiddelde
df <- n - 1 # aantal vrijheidsgraden
alpha <- 0.05
t_crit <- qt(1 - alpha/2, df = df) # kritieke waarde t-verdeling
ondergrens <- m - t_crit*SEM # ondergrens BHI
bovengrens <- m + t_crit*SEM # bovengrens BHI
c(ondergrens, bovengrens)[1] 110.9603 120.8174Het volgende script voert de hele berekening uit:
spanwijdte <- c(117, 106, 123, 111, 124, 120, 108 ,115, 119)
t.test(spanwijdte)$conf.int[1] 110.9603 120.8174
attr(,"conf.level")
[1] 0.95T_m <- c(60.7, 58.3, 60.0, 62.7, 61.7, 60.5, 57.5, 59.1, 59.3, 55.9)
t.test(T_m)$conf.int[1] 58.13292 61.00708
attr(,"conf.level")
[1] 0.95Als een verdeling rechts-scheef is, zijn de kwantielen in de rechterstaart hoger dan verwacht op basis van de normale verdeling. Dan liggen de punten dus boven de diagonale lijn.
Op de \(y\)-as kun je bovendien aflezen wat typische waarden zijn voor de variabele.
Met die twee hints kun je de puzzel oplossen:
Merk op dat de QQ-plot van de variabele SleepHrsNight de vorm heeft van een trappetje. Dat laat zien dat het aantal uren slaap blijkbaar alleen in hele uren kon worden opgegeven. Dat maakt de variabele in feite discreet.
Formule: \[ \mu_X = \overline{X} \pm t_{0{,}05 (2) \mathrm{(n-1)}}\, \mathrm{SE}_{\overline{X}}. \]
Gegeven:
Berekening:
Reken \(\mathrm{SE}_{\overline{X}}\) uit met de formule:
\[\mathrm{SE}_{\overline{X}} = \cfrac{s_X}{\sqrt{n}} = \cfrac{8{,}6\textrm{\,g}}{\sqrt{16}} = 2{,}15\textrm{\,g}.\]
Aantal vrijheidsgraden: \(\mathrm{df}= n - 1 = 15\).
Bereken \(t_{0,05(2)(n-1)}\) met R:
alpha <- 0.05
qt(1 - alpha/2, df = 15)[1] 2.13145Formule invullen:
\[ \mu_X = \overline{X} \pm t_{0{,}05 (2) \mathrm{(n-1)}}\, \mathrm{SE}_{\overline{X}} = (44{,}1 \pm 4{,}6)\textrm{\,g}. \]
Volledig in R:
# gegeven
m <- 44.1 # gemiddelde steekproef
s_X <- 8.6
n <- 16
# SEM berekenen
SEM <- s_X/sqrt(n)
# kritieke t-waarde berekenen
df = n-1
alpha <- 0.05
t_crit <- qt(1 - alpha/2, df = df)
# betrouwbaarheidsinterval berekenen en deze bijvoorbeeld in een vector weergeven:
c( m - t_crit*SEM, m + t_crit*SEM)[1] 39.51738 48.68262Lijkt een normale verdeling bij voorbaat een redelijke optie? Overwegingen:
Al met al lijkt een normaal model bij voorbaat niet erg gepast.
Code:
library("NHANES")
data("NHANES")Code:
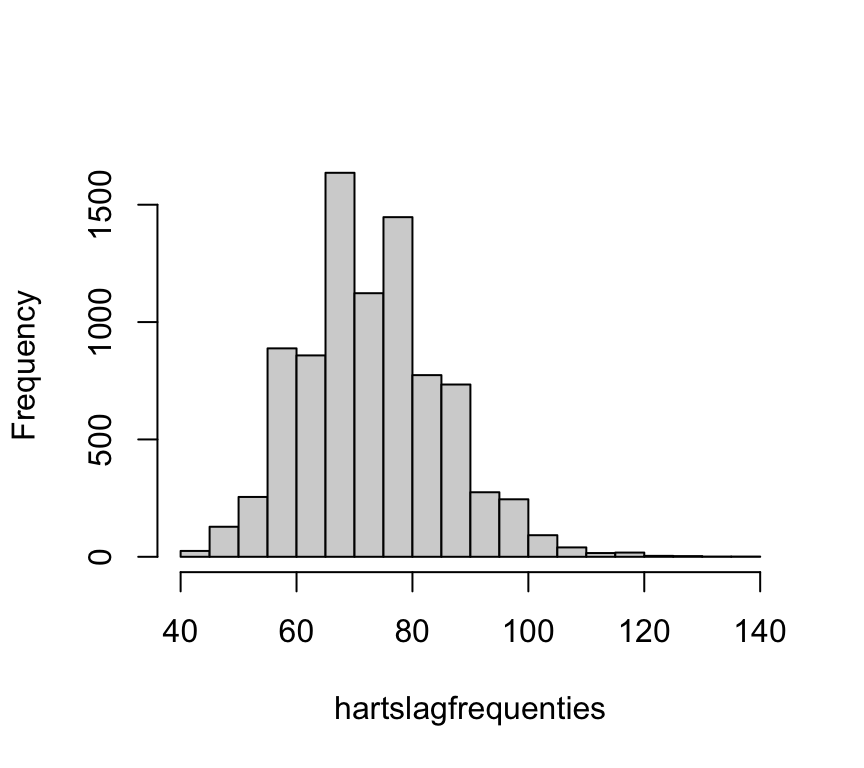
hartslagfrequenties <- na.omit(NHANES$Pulse)
length(hartslagfrequenties)[1] 8563Er blijven na verwijderen van NA’s dus 8563 waarnemingen over. Dat zou je zorgen over de toepasbaarheid van het Normale Model moeten verzachten: bij zo enorm veel gegevens is het model erg robuust.
We maken een histogram en een boxplot:
::: {.cell}
```{.r .cell-code}
hist(hartslagfrequenties, main = NULL)
```
::: {.cell-output-display}
{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
boxplot(hartslagfrequenties)
```
::: {.cell-output-display}
{width=100%}
:::
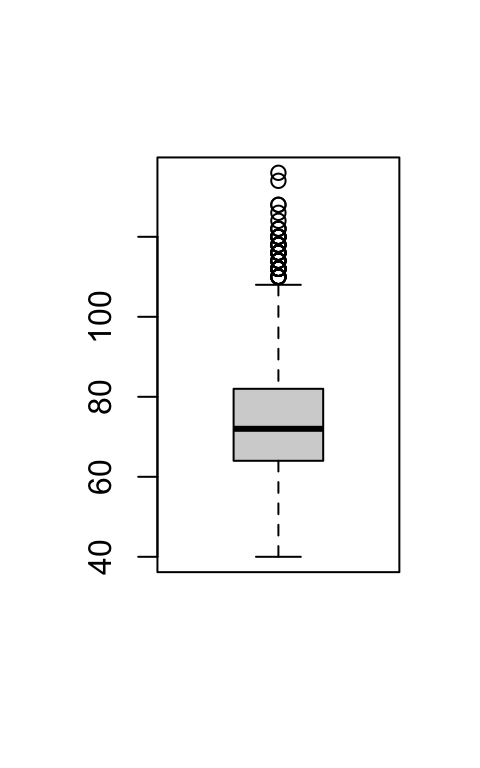
:::Beide plots bevestigen dat de verdeling iets scheef is.
Het histogram ziet er klokvormig uit,
maar heeft een dikkere rechterstaart.
De boxplot laat dat nog duidelijker zien:
de box ziet er symmetrisch uit, maar er zijn veel uitbijters aan de bovenkant.
Verder zien we geen vreemde zaken, zoals frequenties die bij voorbaat
onmogelijk zijn.Code:
mean(hartslagfrequenties)[1] 73.55973median(hartslagfrequenties)[1] 72Of, handig:
summary(hartslagfrequenties) Min. 1st Qu. Median Mean 3rd Qu. Max.
40.00 64.00 72.00 73.56 82.00 136.00 De mediaan is iets kleiner dan het gemiddelde. De afstand van Q1 naar de mediaan (8,00) is ook kleiner dan van de mediaan naar Q3 (10,00). Dat is in lijn met de iets rechts-scheve verdeling.
sd(hartslagfrequenties) # standaarddeviatie[1] 12.15542IQR(hartslagfrequenties) # interkwartielafstand[1] 18De meeste gegevens liggen in het gebied van 64 (Q1) tot 82 (Q3), in lijn met de verwachting.
Tot slot bestuderen we de Normale QQ-plot:
Het midden van de verdeling volgt netjes de kwartielen van de normale verdeling, maar in beide staarten zijn de waargenomen waarden groter dan op basis van de normale verdeling verwacht zou worden. (De punten liggen boven de diagonale lijn.) Dat laat zien dat ten opzichte van de normale verdeling de punten in beide staarten een te hoge waarde hebben. De linkerstaart van de verdeling is dus korter, de rechterstaart juist langer dan bij een normale verdeling.
Code:
resultaat.t.test <- t.test(hartslagfrequenties)
resultaat.t.test$conf.int[1] 73.30224 73.81723
attr(,"conf.level")
[1] 0.95Ja! Hoewel de verdeling duidelijk scheef is (zoals verwacht!) is het aantal datapunten zó groot dat een BHI op basis van het Normale Model prima zou moeten werken. We weten de gemiddelde hartslagfrequentie behoorlijk precies: het ligt met 95% zekerheid ergens tussen 73,3 en 73,8 per minuut.
Er is wel een belangrijke caveat: Er waren behoorlijk veel regels waar NA was ingevuld, en die hebben we verwijderd. De vraag is: waarom is bij deze mensen niets ingevuld? Is er iets bijzonders aan de hand met deze groep? Als dat zo is, dan kan er bias in de overige gegevens zijn geslopen.
Code voor onderdelen a tot f:
library("NHANES")
data("NHANES")
# POPULATIEGEGEVENS
gewichten <- na.omit(NHANES$Weight) # filter NA's weg
populatiegemiddelde <- mean(gewichten) # bereken populatiegemiddelde
populatiegemiddelde # print populatiegemiddelde
# STEEKPROEF
# replace = FALSE zorgt ervoor dat we niet twee keer dezelfde persoon
# in de steekproef kunnen opnemen
steekproefgrootte <- 30
# !!! code vanaf hier herhalen om meerdere steekproeven te proberen
steekproef <- sample(
gewichten, size = steekproefgrootte, replace = FALSE
)
steekproef # steekproef printen
# BHI UITREKENEN
resultaten.t.test <- t.test(steekproef)
bhi <- resultaten.t.test$conf.int
bhi # bhi printen
# Ligt het populatiegemiddelde binnen het BHI?
populatiegemiddelde >= bhi[1] && populatiegemiddelde <= bhi[2]Als je deze code meerdere keren runt, trek je steeds een andere steekproef. In de meeste gevallen zou het populatiegemiddelde in het interval moeten liggen. Om precies te zijn: als de berekening van het BHI voor deze bimodale populatie werkt, zou dat in precies 95% van de gevallen zo moeten zijn. Dat test je in g.
De code voert \(5\cdot 10^4\) steekproeven uit en berekent het percentage van de herhalingen waarbij de populatieparameter in het BHI ligt. Als het goed is, ligt dat percentage erg dicht bij 95%.
De formule voor het BHI werkt dus bij \(n = 30\), ondanks dat de populatie niet normaal verdeeld is.
Pas in de code enkel de steekproefgrootte aan:
steekproefgrootte <- 4 # Grootte van elke steekproefIn alle redelijkheid mogen we nu niet verwachten dat de methode robuust is, want de steekproef is nu wel erg klein. Toch valt de schade mee: het percentage ligt nu rond 95,8%. (Bij \(5\cdot 10^4\) herhalingen kun je er een paar tienden naast zitten.) Dat is nog niet eens zo slecht!
Die kans is gelijk aan de kans om 5 keer achter elkaar “kop” te gooien met een eerlijke munt:
\[\left(\frac{1}{2}\right)^5 = 0{,}031.\]
Een kans van 0,031 is een kleine kans. Het is dus zeker opmerkelijk als alle vijf de keren de jongens in de meerderheid zijn. Dat geeft zeker een indicatie dat er systematisch meer jongetjes dan meisjes worden geboren.
Aan de andere kant, toevallige gebeurtenissen met een kans in de orde van 0,031 zijn nu ook weer niet zó uitzonderlijk. Heb je tijdens een spelletje wel eens drie keer 6 achter elkaar gegooid? Die kans is veel kleiner.
De meest gepaste conclusie is daarom dat de observatie opmerkelijk is, maar dat voor een betrouwbare conclusie gegevens nodig zijn voor een groter aantal jaren. Een goede reden voor vervolgonderzoek.
De nulhypothese is de sceptische hypothese. In dit geval is dat hypothese A, die ervan uitgaat dat het vaccin geen effect heeft. Hypothese B is daarvan de ontkenning en is dus de alternatieve hypothese.
Het bedrijf wil natuurlijk aantonen dat dat hypothese B waar is. (Natuurlijk hoopt het bedrijf bovendien dat de kans op infectie kleiner is voor de vaccin-groep, niet groter!)
Om aan te tonen dat B waar is, probeert men bewijs te verzamelen tegen hypothese A. Net als in de casus van Arbuthnot gebeurt dat door te laten zien dat de waarnemingen zeer onwaarschijnlijk zijn onder de nulhypothese.
\(H_\textrm{0}\): \(p_\textrm{J} = \frac{1}{2}\).
Als er ieder jaar een kans is van \(p_\textrm{J} = \frac{1}{2}\) dat de jongens in de meerderheid zijn, dan verwacht ik dat dat in 82 jaar \(82\times \frac{1}{2} = 41\) keer gebeurt. Dat is de verwachtingswaarde. Inderdaad zie je in het plaatje dat het gemiddelde bij 41 ligt. (De verdeling is symmetrisch, en dus is het gemiddelde gelijk aan de modus en de mediaan.)
Onder \(H_\textrm{0}\) is de \(p_\textrm{J} = \frac{1}{2}\). De kans dat de meisjes in de meerderheid zijn is dus gelijk aan de kans dat de jongetjes dat zijn. Dat betekent dat de kans dat de jongens \(X\) keer in de meerderheid zijn gelijk is aan de kans dat juist de meisjes \(X\) keer in de meerderheid zijn. Daardoor is de kans op uitkomst \(X\) gelijk aan de kans op uitkomst \(82 - X\). Dat maakt de verdeling symmetrisch.
Dat zijn de uitkomsten met \(X \le 34\) en de uitkomsten \(X\ge 48\). Die uitkomsten zijn extremer in de zin dat ze verder in de staarten van de verdeling liggen, en verder van de verwachtingswaarde \(X = 41\).
Dat zijn de staven die paars gekleurd zijn, behalve de staven voor \(X = 35\) en \(X = 47\).
De oppervlakte van de paarse staven is gegeven als 0,22. In Figuur 25.4 kun je aflezen dat
\(\textrm{Pr}\!\left[X = 35\right] = \textrm{Pr}\!\left[X = 47\right] \approx 0{,}037.\)
De \(P\)-waarde voor waarneming \(X = 34\) is dus ongeveer
\(P = 0{,}22 - 2\times 0{,}037 = 0{,}15.\)
De \(P\)-waarde zegt dat onder \(H_\textrm{0}\) een \(X\) die minstens zo extreem is als de waargenomen \(X = 34\) met kans 0,15 gebeurt. Dan is \(X = 34\) niet erg extreem of verrassend te noemen onder \(H_\textrm{0}\). Dit is dus nauwelijks bewijs tegen \(H_\textrm{0}\).
\(P = 0{,}01\) betekent dat, als \(H_\textrm{0}\) waar is, de kans op een toetsingsgrootheid die minstens zo extreem is als de waarde die daadwerkelijk is waargenomen, gelijk is aan 0,01.
Een \(P\)-waarde van 0,01 betekent dat je zo’n extreme waarde van de toetsingsgrootheid maar één op de honderd keer zou verwachten als \(H_\textrm{0}\) waar is. Dat is een behoorlijk kleine kans en het is dus niet zo gek om dat als highly significant (zeer betekenisvol) te bestempelen.
We kunnen aannemen dat er telkens onafhankelijk een kans \(p\) is dat proefpersoon 2 de juiste keuze maakt.
Als de keuzes van proefpersoon 2 niet samenhangen met de kaart die proefpersoon 1 voor zich heeft, dan is \(p = \frac{1}{4}\). (De gok-kans.) Om aan te tonen dat telepathie een rol speelt, moeten we dus laten zien dat \(p\) afwijkt van \(\frac{1}{4}\).
Die parameter is \(p\), de kans op een juiste keuze door proefpersoon 2. In termen van die parameter:
\(H_\textrm{0}: \quad p = \frac{1}{4}\),
\(H_\textrm{A}: \quad p \neq \frac{1}{4}\).
Het resultaat was \(X = 3\) correcte keuzes.
Onder \(H_\textrm{0}\) is de verwachtingswaarde van het aantal correcte keuzes \(n \times p = 3 \times \frac{1}{4} = \frac{3}{4}\). De uitkomsten \(X = 0\), \(X = 1\), en \(X = 2\) zijn daarom allemaal minder extreem dan \(X = 3\). (\(X = 0\) ligt dichter bij de verwachtingswaarde en heeft bovendien een groter kans dan \(X = 3\).)
In dit geval is de \(P\)-waarde dus
\(P = \textrm{Pr}\!\left[X = 3\right] = \left(\frac{1}{4}\right)^3 = \frac{1}{64}.\)
Bij het standaard significantieniveau van \(\alpha = 0{,}05\) is \(P \le \alpha\). Volgens de besluitprocedure zouden we de nulhypothese dus moeten verwerpen.
Deze \(P\)-waarde is redelijk klein, maar ook weer niet extreem klein. Drie keer goed gokken zou opmerkelijk zijn, maar niet onvoorstelbaar. Daar staat tegenover dat bij voorbaat niet waarschijnlijk lijkt dat telepathie mogelijk is. (Wij gaan er bij het tentamen bijvoorbeeld vanuit dat jullie de multiple-choice antwoorden niet via jullie gedachten kunnen uitwisselen…) Er is in ons lichaam geen orgaan bekend dat bedoeld is voor telepathie, en vanuit de natuurkunde is geen “veld” bekend (zoals het elektromagnetisch veld) dat voor de gegevensoverdracht gebruikt zou kunnen worden. Het zou onredelijk zijn om al die kennis uit het raam te gooien vanwege één klein onderzoekje met een \(P\)-waarde van 0,016.
In plaats daarvan is het logischer om de proefpersonen gedurende 3 maanden twee keer per week terug te laten komen voor een serie van 20 pogingen. Als de score na die 560 pogingen nog steeds 100% is, wordt het wél tijd om je wereldbeeld grondig te herzien.
Onder de eerste hypothese is de kans op 4 keer een 6:
\(\left(\frac{1}{6}\right)^4 = 7{,}7\times 10^{-4}\).
Onder de tweede hypothese is de kans op 4 keer een 6:
\(\frac{1}{2}^4 = 0{,}0625\).
Nee. De uitkomst “4 keer een 6” is onder beide hypotheses mogelijk, en dus kunnen we niet met zekerheid zeggen welke waar is.
Onder de eerste hypothese is de uitkomst heel erg onwaarschijnlijk; 81 keer zo onwaarschijnlijk als onder de tweede hypothese. Mijn advies is om je geld in te zetten op hypothese 2.
Deze opgave laat iets belangrijks zien: Als twee alternatieve hypotheses beide niet kunnen worden uitgesloten, wil dat niet zeggen dat ze even geloofwaardig zijn.
Kies het kansmodel: het Normale Model.
Formuleer de hypotheses:
\(H_\textrm{0}:\qquad \mu_X = 52\textrm{\,g}\),
\(H_\textrm{A}:\qquad \mu_X \neq 52\textrm{\,g}\).
Kies de toetsingsgrootheid:
\(t = \cfrac{\overline{X} - 52\textrm{\,g}}{\mathrm{SE}_{\overline{X}}}\).
Verdeling van \(t\) onder \(H_\textrm{0}\):
De \(t\)-verdeling met \(\mathrm{df}= n - 1 = 3\).
Bereken de toetsingsgrootheid op basis van de steekproef.
Achtereenvolgens:
De resultaten hierboven zijn afgerond; maar zorg ervoor dat je altijd met de onafgeronde tussenresultaten doorrekent.
Bereken de \(P\)-waarde: Gebruik R:
P <- 2*pt(-abs(t), df = n - 1)
P[1] 0.175857Conclusies: De \(P\)-waarde is 0,18. Dat is geen opvallend kleine waarde; de waargenomen afwijking van \(\overline{X}\) van \(\mu_0\) verwacht je onder \(H_\textrm{0}\) bij bijna 1 op de 5 steekproeven van deze grootte. Deze gegevens geven dus geen aanleiding om de claim van het bedrijf in twijfel te trekken.
De steekproef was ook wel erg klein; daardoor is de onzekerheid in \(\mu_X\) behoorlijk groot. Met een grotere steekproef kunnen we de standaardfout \(\mathrm{SE}_{\overline{X}}\) omlaag brengen. Het zou kunnen dat we dan wél een statistisch significante afwijking vinden.
De uitvoer is:
One Sample t-test
data: temperaturen
t = -3.0877, df = 9, p-value = 0.01298
alternative hypothesis: true mean is not equal to 37
95 percent confidence interval:
36.13368 36.86632
sample estimates:
mean of x
36.5 Helemaal onderaan staat “mean of x”, met waarde 36,5 (in \(\degree\)C).
Deze drie waarden staan op dezelfde regel:
t = -3.0877, df = 9, p-value = 0.01298Na afronden komen deze resultaten precies overeen met onze eigen berekeningen.
Het BHI loopt van 36,13\(\degree\)C tot 36,87\(\degree\)C. Je kunt dit ook schrijven als \(\mu_X = (36{,}50\pm 0{,}37)\degree\)C.
Attaching package: 'palmerpenguins'The following objects are masked from 'package:datasets':
penguins, penguins_rawDe uitvoer is:
Two Sample t-test
data: snavel_Chinstrap and snavel_Gentoo
t = 2.7694, df = 189, p-value = 0.006176
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
0.3823625 2.2755285
sample estimates:
mean of x mean of y
48.83382 47.50488 Lees af: \(\mathrm{df}= 189\). Er waren metingen voor 68 Chinstrap-pinguins en 123 Gentoo-pinguins. Het juiste aantal is \(n_1 + n_2 - 2 = 68 + 123 - 2 = 189\). R heeft dus het juiste aantal vrijheidsgraden gebruikt.
Het interval is \((0,38\textrm{\,mm}; 2,28\textrm{\,mm})\). Dit is het 95%-BHI van het verschil in de gemiddelden tussen beide populaties, dus \(\mu_1\) - \(\mu_2\).
Het getal 0 ligt niet binnen het BHI. Dat vertelt ons dat de \(P\)-waarde kleiner is dan 0,05.
De \(P\)-waarde van de gewone \(t\)-toets is 0,0062, die van de test van Welch is met 0,0077 iets groter, zoals verwacht.
Hoewel er dus een klein verschil te zien is, verschilt de interpretatie nauwelijks: in beide gevallen is het bewijs sterk dat er een verschil is in de gemiddelde snavellengte tussen beide soorten. Het heeft dus geen serieus nadeel om de test van Welch te gebruiken, terwijl die wel robuuster is voor afwijkingen van de standaarddeviaties.
Omdat de test van Welch minder aannames maakt, is er meer onzekerheid in de schatting. Daarom verwachten we een breder BHI. Dat klopt:
95%-BHI voor de standaard toets: \((0,38\textrm{\,mm}; 2,28\textrm{\,mm})\).
95%-BHI voor de toets van Welch: \((0,36\textrm{\,mm}; 2,30\textrm{\,mm})\).
We willen een hypothese toetsen over het gemiddelde van de populatie; dat is de taak van de \(t\)-toets voor één steekproef.
\(H_\textrm{0}:\qquad \mu_X = 16{,}5,\)
\(H_\textrm{A}:\qquad \mu_X \neq 16{,}5.\)
We importeren de dataset:
massa_manacus <- read.csv(
"https://tbb.bio.uu.nl/bvd/bms/slides/3_Statistiek_data/massa_manacus.csv"
)Welke variabelen? Gebruik str():
str(massa_manacus)'data.frame': 641 obs. of 7 variables:
$ RecordType : chr "Preserved specimen" "Preserved specimen" "Preserved specimen" "Preserved specimen" ...
$ Sex : chr "Female" "Female" "Female" "Female" ...
$ Weight : num 14.5 16 20 14.2 17.2 16 16.4 16.4 18.4 16.1 ...
$ ReferenceW : num 16.7 16.7 16.7 16.7 16.7 16.7 16.7 16.7 16.7 16.7 ...
$ DifferenceW.RefW: num -2.2 -0.7 3.3 -2.5 0.5 -0.7 -0.3 -0.3 1.7 -0.6 ...
$ Latitude : num 5.25 11.32 -3.43 4.57 5.44 ...
$ Logitude : num -68.5 -74.1 -70.1 -71.3 -75.8 ...Aantal datapunten? Vraag het aantal regels op:
nrow(massa_manacus)[1] 641Dit is duidelijk een erg grote dataset.
We zijn geïnteresseerd in de variabele Weight.
Ligging:
summary(massa_manacus$Weight) Min. 1st Qu. Median Mean 3rd Qu. Max.
11.40 15.00 16.30 16.28 17.50 21.00 De mediaan en het gemiddelde zijn bijna gelijk, en ietsje kleiner dan \(\mu_0 = 16{,}5\). De vraag is of dat kleine verschil statistisch significant is.
Spreiding:
sd(massa_manacus$Weight)[1] 1.787385IQR(massa_manacus$Weight)[1] 2.5Visualiseren:
hist(massa_manacus$Weight)
boxplot(massa_manacus$Weight)Beide plots laten een symmetrische, klokvormige verdeling zien, zonder opvallende uitbijters.
We passen het Normale Model toe op de populatie en de steekproef. We nemen dus onder andere aan dat gewicht in de populatie normaal verdeeld is.
Bij voorbaat is het niet vreemd om lichaamsgewicht te modelleren met een normale verdeling, omdat dat een continue variabele is die vaak een klokvormige verdeling heeft.
Een probleem zou kunnen zijn dat een lichaamsgewicht niet negatief kan worden, maar zolang de ligging van de verdeling ver genoeg van 0 afligt is het ontbreken van die staart niet zo relevant.
Er is in deze opgave niet veel informatie gegeven over de manier waarop de steekproef heeft plaatsgevonden. We kunnen dus niet goed beoordelen of de vogels bij benadering aselect geselecteerd zijn. Dat is iets om in gedachten te houden; er kan bias bestaan in deze steekproef.
QQ-plot:
De QQ-plot laat zien dat de verdeling grotendeels in lijn is met de kwantielen van de normale verdeling.
Alles overziend kan de verdeling goed beschreven worden met de normale verdeling. Gezien de grootte van de dataset zouden zelfs veel grotere afwijkingen geen probleem moeten zijn.
De \(t\)-toets uitvoeren:
t.test(massa_manacus$Weight, mu = 16.5)
One Sample t-test
data: massa_manacus$Weight
t = -3.1384, df = 640, p-value = 0.001777
alternative hypothesis: true mean is not equal to 16.5
95 percent confidence interval:
16.13981 16.41707
sample estimates:
mean of x
16.27844 Het 95%-BHI is \((16{,}14\textrm{\,g}; 16{,}42\textrm{\,g})\). We zijn dus 95% zeker dat het ware gemiddelde in dat interval ligt.
Omdat \(16,5\textrm{\,g}\) niet in het 95%-BHI ligt, kunnen we ook al concluderen dat \(P < 0{,}05\).
De \(P\)-waarde 0,0018. Dat is sterk bewijs tegen de nulhypothese. (Als \(H_\textrm{0}\) waar is, krijg je zo’n grote afwijking maar eens in de ~500 steekproeven!)
De BHI geeft je een schatting van het lichaamsgewicht van deze vogels, plus je onzekerheid daarin. Je kunt daarmee dus ook inschatten hoe ver de werkelijkheid van \(\mu_0 = 16{,}5\)g afligt. Met een geschat gemiddelde van \(16{,}3\)g is het verschil eigenlijk maar klein.
De \(P\)-waarde vertelt je precies hoe onwaarschijnlijk het waargenomen steekproefgemiddelde is onder \(H_\textrm{0}\), en dus hoe zwaarwegend dit bewijs is tegen \(H_\textrm{0}\). Het 95%-BHI vertelt ons dat \(P < 0,05\), maar niet hoe groot \(P\) precies is.
We willen de gemiddelden van twee populaties vergelijken; we hebben dus een \(t\)-toets voor twee onafhankelijke steekproeven nodig
\(H_\textrm{0}:\qquad \mu_1 = \mu_2,\)
\(H_\textrm{A}:\qquad \mu_1 \neq \mu_2.\)
Code:
man <- massa_manacus$Weight[massa_manacus$Sex == "Male"]
vrouw <- massa_manacus$Weight[massa_manacus$Sex == "Female"]Code:
length(man)[1] 201length(vrouw)[1] 252Mannetjes:
::: {.cell}
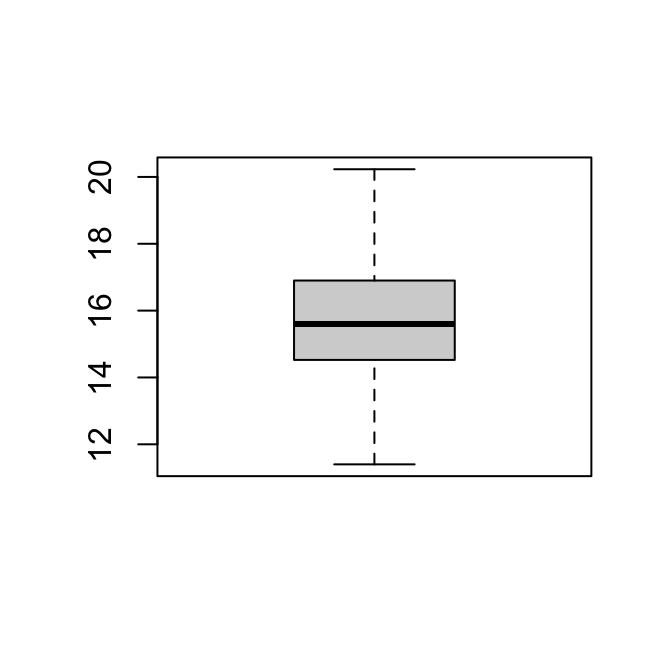
```{.r .cell-code}
boxplot(man)
```
::: {.cell-output-display}
{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
hist(man)
```
::: {.cell-output-display}
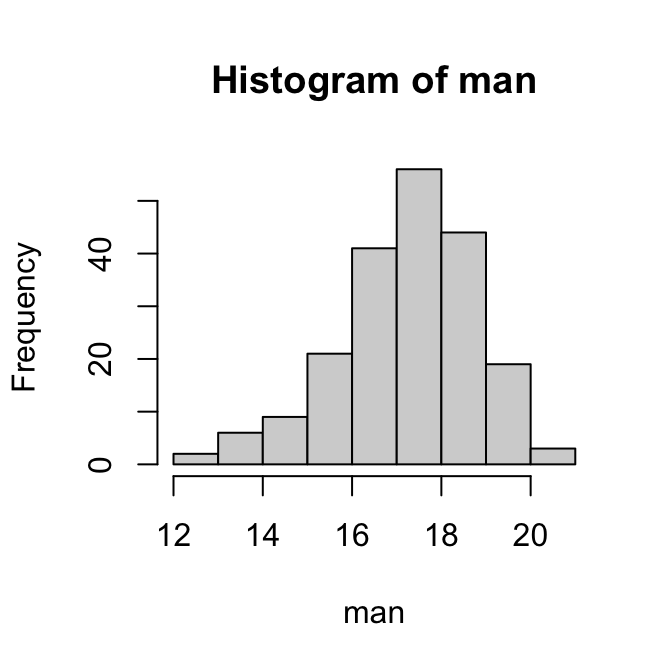{width=100%}
:::
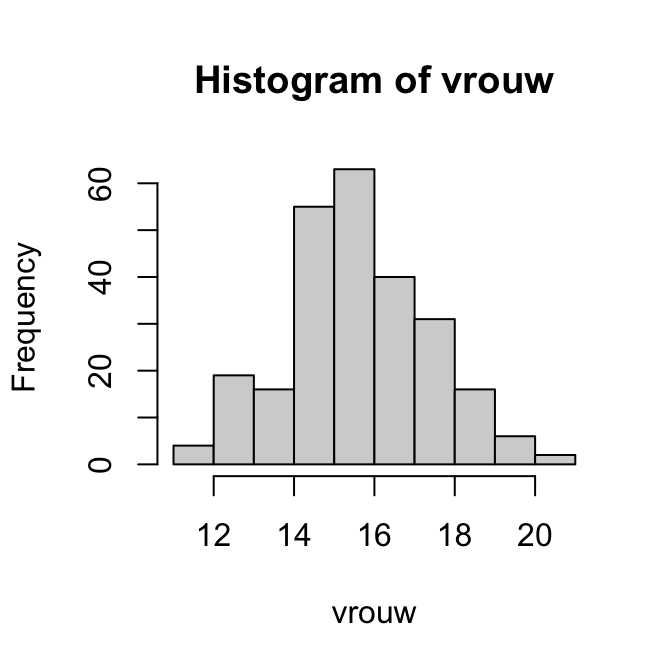
:::**Vrouwtjes:**::: {.cell}
```{.r .cell-code}
boxplot(vrouw)
```
::: {.cell-output-display}
{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
hist(vrouw)
```
::: {.cell-output-display}
{width=100%}
:::
:::**Ligging:**
::: {.cell}
```{.r .cell-code}
summary(man)
```
::: {.cell-output .cell-output-stdout}
```
Min. 1st Qu. Median Mean 3rd Qu. Max.
12.70 16.60 17.50 17.38 18.44 21.00
```
:::
```{.r .cell-code}
summary(vrouw)
```
::: {.cell-output .cell-output-stdout}
```
Min. 1st Qu. Median Mean 3rd Qu. Max.
11.40 14.54 15.60 15.70 16.90 20.23
```
:::
:::
De mannetjes lijken net iets groter dan de vrouwtjes,
al moet de toets uitwijzen of dat statistisch significant is.
**Spreiding:**
::: {.cell}
```{.r .cell-code}
sd(man)
```
::: {.cell-output .cell-output-stdout}
```
[1] 1.568567
```
:::
```{.r .cell-code}
IQR(man)
```
::: {.cell-output .cell-output-stdout}
```
[1] 1.84
```
:::
:::
::: {.cell}
```{.r .cell-code}
sd(vrouw)
```
::: {.cell-output .cell-output-stdout}
```
[1] 1.807133
```
:::
```{.r .cell-code}
IQR(vrouw)
```
::: {.cell-output .cell-output-stdout}
```
[1] 2.3625
```
:::
:::We nemen aan dat het Normale Model van toepassing is op beide populaties. Bovendien nemen we aan dat de standaarddeviatie van beide populaties gelijk is.
Bij voorbaat lijkt het, net als in de vorige opgave, niet gek op lichaamsgewicht te modelleren met een normale verdeling. Bij voorbaat zou je ook niet verwachten dat de standaarddeviatie sterk verschilt tussen mannelijke en vrouwelijke vogels. Het is dus niet bij voorbaat gek om het Normale Model te gebruiken.
Kijkend naar de maten van spreiding hierboven lijken de vrouwtjes een iets grotere spreiding te hebben dan de mannetjes. Maar het verschil is niet groot, en bij dit grote aantal waarnemingen zou dat geen probleem moeten zijn.
Om de normaliteit van de gegevens te beoordelen, kijken we naar de histogrammen en boxplots hierboven. De verdeling van de vrouwtjes ziet er redelijk symmetrisch uit; die van de mannetjes is een beetje links-scheef.
Om dat nog iets beter te beoordelen, maken we QQ-plots:
::: {.cell}
```{.r .cell-code}
qqnorm(man)
qqline(man)
```
::: {.cell-output-display}
{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
qqnorm(vrouw)
qqline(vrouw)
```
::: {.cell-output-display}
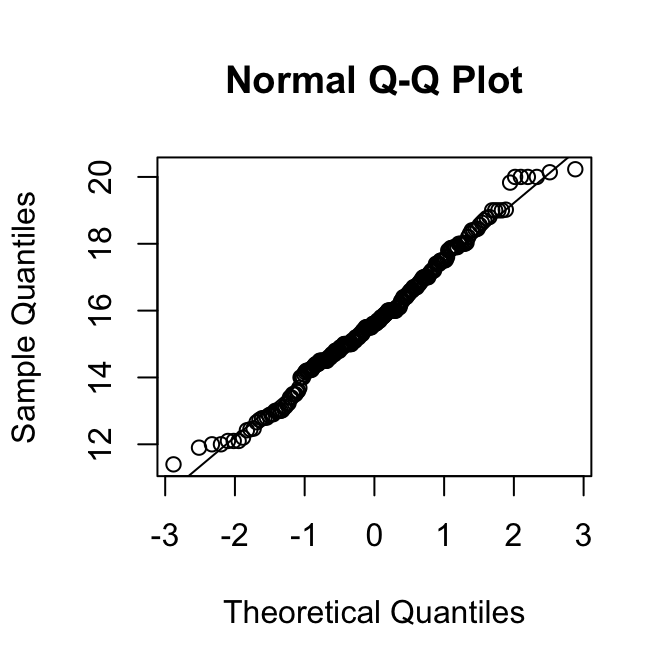{width=100%}
:::
:::We zien weer dat de verdeling van de mannetjes wat links-scheef is,
maar die van de vrouwtjes niet.
Gezien de grootte van deze steekproeven,
is de kleine scheefheid van de mannetjes geen groot probleem.Code:
t.test(man, vrouw, var.equal = TRUE)
Two Sample t-test
data: man and vrouw
t = 10.417, df = 451, p-value < 2.2e-16
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
1.363085 1.997010
sample estimates:
mean of x mean of y
17.38279 15.70274 Het 95%-BHI van het verschil tussen de twee gemiddelden is \((1{,}36\textrm{\,g}; 2{,}00\textrm{\,g})\). De mannetjes zijn een tikje zwaarder.
De \(P\)-waarde is kleiner dan \(2{,}2\times 10^{-16}\). (\(P\)-waardes kleiner dan \(2,2\times 10^{-16}\) kan R niet precies berekenen.) Dit is natuurlijk een heel erg kleine \(P\)-waarde; dit is dus heel erg sterk bewijs tegen de nulhypothese.
Het BHI vertelt je dat het verschil op zich niet groot is: maximaal een gram of 2. Dat kun je aan de superkleine \(P\)-waarde niet aflezen.
Andersom kan je uit het BHI enkel afleiden dat \(P<0{,}05\). Dat het bewijs zo sterk is, kun je daaraan dus niet zien.
Er zijn twee steekproeven: afzetting aan de oostkant en afzetting aan de westkant. Maar, deze zijn duidelijk gepaard, want van iedere boom is de afzetting aan beide kanten gemeten. De kurkafzetting zal per boom verschillen; grote bomen zullen misschien aan alle kanten een dikkere kurklaag hebben dan kleine bomen. Daarom willen we de bomen niet onderling vergelijken, maar onderzoeken of er een systematisch verschil is tussen de oost- en west-kant van dezelfde boom. We willen dus een \(t\)-toets voor gepaarde gegevens uitvoeren.
\(H_\textrm{0}:\qquad \mu_d = 0,\)
\(H_\textrm{A}:\qquad \mu_d \neq 0.\)
Code:
kurk <- read.csv(
"https://tbb.bio.uu.nl/bvd/bms/slides/3_Statistiek_data//kurk.csv"
)Voer uit:
str(kurk)'data.frame': 28 obs. of 4 variables:
$ NOORD: int 72 60 56 41 32 30 39 42 37 33 ...
$ OOST : int 66 53 57 29 32 35 39 43 40 29 ...
$ ZUID : int 76 66 64 36 35 34 31 31 31 27 ...
$ WEST : int 77 63 58 38 36 26 27 25 25 36 ...head(kurk) NOORD OOST ZUID WEST
1 72 66 76 77
2 60 53 66 63
3 56 57 64 58
4 41 29 36 38
5 32 32 35 36
6 30 35 34 26De uitvoer van str() laat zien dat er 4 variabelen zijn, en dus 4 kolommen.
De uitvoer str() geeft aan dat er 28 obs.(observations) zijn. Er zijn dus ook 28 rijen.
De uitvoer van str() laat zien dat elke kolom int (integer) als datatype heeft.
De gegevens zijn, uitgedrukt in mm, gehele getallen, dus het is niet gek dat R ze als integers interpreteert. Maar, eigenlijk is de kurkdikte een continue variabele, dus numeric zou beter passen. Het is netjes om de gegevens om te zetten, maar de meeste functies zetten integers vanzelf om naar numeric als de berekening daarom vraagt, dus essentieel is het niet.
kurk$WEST <- as.numeric(kurk$WEST)
kurk$OOST <- as.numeric(kurk$OOST)Eerst de boxplots:
::: {.cell}
```{.r .cell-code}
boxplot(kurk$OOST, main = "OOST")
```
::: {.cell-output-display}
{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
boxplot(kurk$WEST, main = "WEST")
```
::: {.cell-output-display}
{width=100%}
:::
:::Nu de histogrammen:::: {.cell}
```{.r .cell-code}
hist(kurk$OOST, main = "OOST")
```
::: {.cell-output-display}
{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
hist(kurk$WEST, main = "WEST")
```
::: {.cell-output-display}
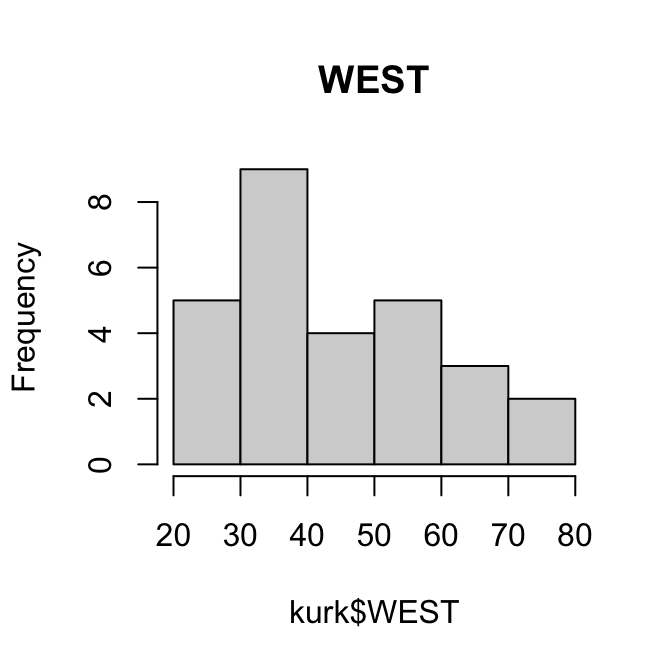{width=100%}
:::
:::Zowel de boxplot als de histogrammen laten zien
dat de verdelingen lichtelijk rechts-scheef zijn.
Ook valt op dat er flinke variatie is in de afzetting,
want die varieert van 20 tot 80 mm.
Verder zijn er geen vreemde punten of uitbijters.Ligging:
summary(kurk$OOST) Min. 1st Qu. Median Mean 3rd Qu. Max.
29.00 35.00 41.50 46.18 54.25 80.00 summary(kurk$WEST) Min. 1st Qu. Median Mean 3rd Qu. Max.
25.00 35.00 41.50 45.18 58.00 77.00 De mediaan van OOST en WEST zijn gelijk; er is wel een klein verschil in gemiddelde.
Spreiding:
sd(kurk$OOST)[1] 14.83003IQR(kurk$OOST)[1] 19.25sd(kurk$WEST)[1] 15.03343IQR(kurk$WEST)[1] 23De spreiding van beide variabelen is vergelijkbaar, al lijken de waarden van WEST iets meer spreiding te hebben dan die van OOST.
Het statistische model veronderstelt dat de de verschillen tussen OOST en WEST binnen de paren voldoen aan het Normale Model.
Bij voorbaat zou het goed kunnen dat die verschillen een belvormige verdeling hebben, en als de bomen aselect zijn gekozen, dan zouden deze verschillen ook onafhankelijk moeten zijn. Het normale model kan dus een redelijke keuze zijn.
Om te bekijken of de gegevens zelf aanleiding geven tot twijfel maken we een histogram en een QQ-plot. Let op! De aanname gaat over de verschillen, en dus moeten we eerst de verschillen uitrekenen en dan plotjes maken op basis van die verschillen:
::: {.cell}
```{.r .cell-code}
verschillen <- kurk$OOST - kurk$WEST
hist(verschillen)
```
::: {.cell-output-display}
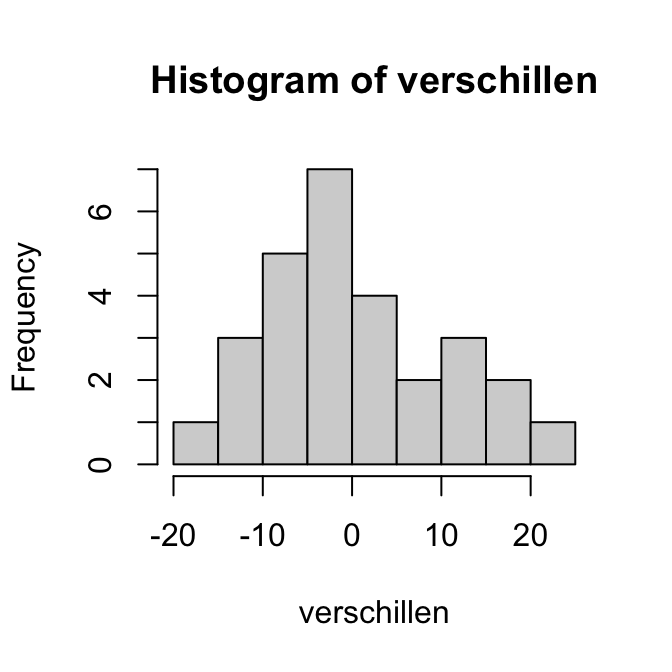{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
qqnorm(verschillen)
qqline(verschillen)
```
::: {.cell-output-display}
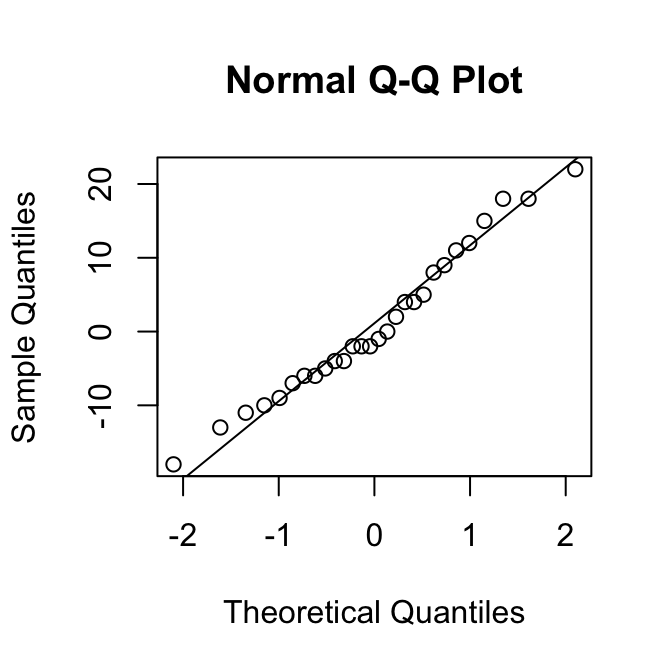{width=100%}
:::
:::Het histogram ziet er niet perfect normaal verdeeld uit.
Maar, het aantal waarnemingen is niet erg groot,
en dus zijn er per klasse maar weinig waarnemingen in het histogram.
Dan ziet het histogram er al snel rommelig uit.
De QQ-plot ziet er niet heel verkeerd uit.
Hoewel we niet goed kunnen beoordelen of de verschillen dus
in de populatie normaal verdeeld zijn,
zien we ook geen duidelijke afwijkingen.
Met die caveats kunnen we het model dus gebruiken.Code:
t.test(kurk$OOST, kurk$WEST, paired = TRUE)
Paired t-test
data: kurk$OOST and kurk$WEST
t = 0.52092, df = 27, p-value = 0.6067
alternative hypothesis: true mean difference is not equal to 0
95 percent confidence interval:
-2.938865 4.938865
sample estimates:
mean difference
1 Het 95%-BHI: \((-2{,}94\textrm{\,mm}; 4{,}94\textrm{\,mm})\). Het ware gemiddelde verschil tussen OOST en WEST in de populatie ligt dus met 95% zekerheid in dit interval.
Omdat 0mm in dit interval ligt, weten we al dat de \(P\)-waarde groter dan 0,05 moet zijn.
We vinden \(P=0{,}61\). Dat is verre van significant. Deze gegevens vormen dus geen bewijs tegen de nulhypothese.
Het 95%-BHI vertelt ons dat het ware gemiddelde verschil tussen OOST en WEST nog best -2,5mm of 4,9mm kan zijn. We hebben dus geen significant bewijs gevonden voor een verschil, maar kunnen tegelijkertijd niet uitsluiten dat er een klein verschil is.
Uit de BHI kon worden opgemaakt dat \(P>0{,}05\), maar niet hoe groot de \(P\)-waarde precies is. De exacte \(P\)-waarde van 0,61 geeft extra informatie, die geeft geen enkele suggestie van een verschil.
Het is moeilijk hiervoor een antwoordmodel te geven, want iedere student heeft andere parameters geprobeerd en andere resultaten gevonden.
Duidelijk is wel dat je de \(t\)-toets voor twee onafhankelijke steekproeven moet uitvoeren. Als de standaarddeviaties van de twee condities die je wilt vergelijken sterk verschillen, kun je ook voor de toets van Welch kiezen.
Zoals altijd bekijk je vooraf of het Normale Model van toepassing lijkt! Je script zal er dan ongeveer zo uit zien:
aapjes1 <- c( ) # jouw resultaten hier!
aapjes2 <- c( ) # jouw resultaten hier!
# resultaten bekijken en vergelijken
boxplot(aapjes1)
hist(aapjes1)
boxplot(aapjes2)
hist(aapjes2)
summary(aapjes1)
summary(aapjes2)
sd(aapjes1)
IQR(aapjes1)
sd(aapjes2)
IQR(aapjes2)
qqnorm(aapjes1)
qqline(aapjes1)
qqnorm(aapjes2)
qqline(aapjes2)
t.test(aapjes1, aapjes2, var.equal = TRUE) # of FALSE voor WelchAls je twee flink verschillende condities vergelijkt, zoals eetsnelheid 0,5 en eetsnelheid 5, dan zul je bij \(n = 10\) herhalingen per conditie waarschijnlijk een statistisch significant verschil vinden. Kies je de condities dicht bij elkaar, zoals 0,5 en 0,6, dan is de kans groot dat je geen significant bewijs vindt voor een verschil.
Transcriptiefactoren zijn eiwitten (of eiwitcomplexen) die de expressie van andere genen reguleren. Als je een associatie vindt tussen de expressie van een transcriptiefactor en die van een gen, ligt het dus voor de hand dat de causaliteit van de expressie van de transcriptiefactor naar de expressie van het gen loopt. Dat is diagram (a).
Hoewel diagram (a) voor de hand ligt, moet je erg uitkijken met het trekken van conclusies, want ook diagram (b) en (c) kunnen het geval zijn.
Bij diagram (b) loopt het causale verband de andere kant op. Dat zou natuurlijk kunnen als gen X zelf ook codeert voor een transcriptiefactor. Een andere mogelijkheid is dat X het gen is voor een metabool enzym dat een metaboliet produceert die de expressie van TF induceert. De causale pijl kán dus ook de andere kant op gericht zijn.
Bij diagram (c) raken de expressies van X en TF geassocieerd doordat ze beiden beïnvloed worden door een andere variabele. Dat zou bijvoorbeeld een andere transcriptiefactor kunnen zijn die zowel X als TF reguleert.
Kortom: wees voorzichtig met conclusies over causaliteit. Hoewel het causale diagram (a) voor de hand ligt, kan het in werkelijkheid anders zijn.
Zowel lichaamslengte als IQ worden beïnvloed door omstandigheden tijdens de ontwikkeling van het kind. Een goede voeding, slaap, gezondheid, en een veilige omgeving dragen allemaal positief bij aan lichaamslengte én IQ. Dat kunnen dus allemaal confounders zijn.
Afhankelijk van de confounder die je hebt gekozen, zal het diagram er ongeveer zo uitzien:
graph TD; A[Leefomstandigheden tijdens ontwikkeling] --> B[Lichaamslengte] A --> C[IQ]
In dit diagram hebben Roken en Alcoholgebruik één confounder: Genetische aanleg.
Er is in dit diagram geen variabele die (direct of indirect) zowel Roken als Lichaamsbeweging beïnvloedt. Er is dus geen confounder.
Roken heeft direct effect op Hartproblemen en Lichaamsbeweging en is dus een confounder. Socio-economische status beïnvloedt Lichaamsbeweging, en indirect ook Hartproblemen, via Alcoholgebruik. Dat kan dus ook een confounder zijn.
Er wijzen twee pijlen naar Alcoholgebruik: vanuit Genetische aanleg en vanuit Socio-economische status. Alcoholgebruik is dus een collider.
Als we een groep selecteren op basis van Alcoholgebruik, dan kan binnen die groep dus een associatie ontstaan tussen Socio-economische status en Genetische aanleg die er in de volledige populatie niet is. Dat is collider bias.
Maar daar stoppen de problemen niet! Omdat Genetische aanleg ook effect heeft op Roken, kan er nu ook een valse associatie gevonden worden tussen Socio-economische status en Roken. Op dezelfde manier kan de collider bias de associatie tussen Genetische aanleg en Lichaamsbeweging verstoren.
In dit geval leidt de selectie van een groep op basis van Alcoholgebruik dus tot verstoringen in de associaties tussen vrijwel alle variabelen.
We zoeken naar variabelen die, direct of indirect, een effect hebben op zowel Gewicht moeder voor de zwangerschap als Keizersnede. Voorbeelden zijn:
Leeftijd moederChronische hoge bloeddrukLichaamslengte moederRas moederHet geboortegetal daalde in de periode 1965–1980 stevig. Dat zorgt voor vergrijzing van de populatie, net als in Nederland, met allerlei nadelige gevolgen.
De data geven duidelijk de oorzaak van het probleem weer: de afname van het aantal ooievaars. Want “ieder kind weet”: geen ooievaars, geen baby’s!
Er moet duidelijk geïnvesteerd worden in betere omstandigheden voor ooievaars. Hoe dat precies moet, vertelt Prof. Sies hier niet, maar je zou kunnen denken aan voldoende nestpalen.
De tentamens anonimiseren, zodat de docent tijdens het beoordelen niet kan weten van wie het tentamen is en waarnemersbias dus geen kans krijgt. Binnen software als Remindo en Blackboard kan anoniem nakijken worden ingesteld.
Een persoon buiten de selectiecommissie kan alle ingestuurde documenten langsgaan en informatie waaruit gender of etniciteit kan worden afgeleid onleesbaar maken (“zwartlakken”).
Audio-experts maken hiervoor gebruik van geblindeerde A-B-testen. Je vraagt dan iemand om in random volgorde gewone kabels (conditie A) of de dure kabels (conditie B) aan te sluiten, zonder dat jij dat kunt zien; jij luistert naar de muziek en probeert te horen of je naar conditie A of conditie B luistert. Als je consistent het juiste antwoord weet, kun je het verschil blijkbaar horen.
Dat valt meestal vies tegen.
De doorstroomtoets heeft verschillende nadelen: het is een momentopname, toetst slechts een beperkt aantal kennisgebieden en vaardigheden, en kan scholen onbedoeld aansporen om vooral te trainen op een hoge toetsscore, in plaats van breed onderwijs aan te bieden. Leraren hebben vaak een veel genuanceerder beeld van hun leerlingen dan een enkele toets ooit kan vastleggen. Om die reden pleiten sommigen voor het afschaffen van eindtoetsen.
Maar het is ook overtuigend aangetoond dat bepaalde groepen leerlingen structureel door hun leraren worden onderschat. Een belangrijk voordeel van de doorstroomtoets is daarom dat deze blind beoordeelt: twee leerlingen met dezelfde score krijgen hetzelfde advies, ongeacht hun achtergrond of uiterlijk. Zo biedt de toets een objectief extra datapunt waarmee waarnemersbias kan worden tegengegaan.
Het antwoord moet natuurlijk regression to the mean zijn.
De prestaties van een piloot hangen natuurlijk af van diens vaardigheid, maar schommelen ook van dag tot dag. Instructeurs zullen geneigd zijn een piloot te complimenteren wanneer deze een bijzonder goede prestatie levert. Juist omdat complimenten vooral gegeven worden bij bovengemiddelde prestaties, is de kans groot dat de volgende poging minder goed uitvalt. Dat kan de indruk wekken dat het compliment de oorzaak was van de achteruitgang in prestatie, maar dat hoeft niet zo te zijn: ook zonder compliment zou de volgende poging waarschijnlijk minder goed zijn geweest.
Voor bestraffen geldt hetzelfde, maar dan omgekeerd: de instructeur zal een piloot vooral berispen wanneer die opvallend slecht presteert. Ook zonder die berisping is de kans groot dat de daaropvolgende prestatie beter uitvalt—meer in lijn met het gebruikelijke niveau van de piloot.
Het is dus goed mogelijk dat beloningen wél beter werken dan straffen, maar dat het in de praktijk soms anders lijkt omdat beloningen worden gegeven na een bovengemiddelde prestatie en afstraffingen juist na een ondermaatse prestatie.
Het is niet gek als je schatting er, zeg, 0,1 naast zit. Maar probeer je oog met deze voorbeelden te trainen.
De puntenwolk van Galton heeft ongeveer dezelfde vorm als die van \(r = 0{,}51\) in Figuur 28.3. De correlatiecoëfficiënt moet dus dicht bij die waarde liggen. Later in het hoofdstuk berekenen we de waarde exact.
De antwoorden zijn:
(a) \(r = 0{,}85\)
(b) \(r = 0{,}90\)
(c) \(r = -0{,}53\)
(d) \(r = 0{,}64\)
(e) \(r = 0{,}32\)
(f) \(r = 0{,}03\)
(g) \(r = 0{,}46\)
(h) \(r = -0{,}79\)
We vullen in de formule Vergelijking 28.1 voor \(Y\) ook \(X\) in. Dat levert op:
\[ \begin{align} C_{XX} &= \frac{\sum_{i=1}^{n} (x_i - \bar{x})(x_i - \bar{x})}{n-1}\\ &= \frac{\sum_{i=1}^{n} (x_i - \bar{x})^2}{n-1}. \end{align} \tag{4}\]
Vergelijk Vergelijking 4 met Vergelijking 20.4; dan wordt duidelijk dat
\[ C_{XX} = V_X. \]
De covariantie van een variabele met zichzelf is dus de variantie van die variabele!
De covariantie meet de lineaire samenhang van twee variabelen. Iedere variabele hangt perfect lineair samen met zichzelf. De covariantie van een variabele met zichzelf moet dus wel positief zijn.
We vullen in formule Vergelijking 28.2 voor \(Y\) weer \(X\) in:
\[ r = \frac{C_{XY}} {s_X s_Y} = \frac{C_{XX}}{s_X s_X}. \] In Oefening 28.2 heb je afgeleid dat \(C_{XX} = V_X\), en je wist al dat \(s_X s_X = s_X^2 = V_X\). Invullen levert op:
\[ r = \frac{C_{XX}}{s_X s_X} = \frac{V_X}{V_X} = 1. \] De correlatiecoëfficiënt van een variabele met zichzelf is dus altijd 1.
De puntenwolk van Figuur 28.1 ziet er mooi ovaal uit. Die algemene vorm is consistent met een bivariate normale verdeling.
Wel valt op dat de punten soms horizontaal of verticaal uitlijnen. Dat komt gedeeltelijk doordat lengtes in de dataset oorspronkelijk door Galton in inches zijn gemeten, met een resolutie van \(\frac{1}{4}\) inch. De data die hier gebruikt zijn komen uit de HistData library, waar ze zijn ze omgerekend naar cm en afgerond. Daardoor komen sommige waarden meerdere keren exact voor.
Er is nog een reden voor het uitlijnen van sommige waarden, maar daar komen we zo op.
De verdeling van de ouders is klokvormig, maar lijkt iets te dikke staarten te hebben, en heeft een opvallende piek bij 175cm. De afwijkingen zijn niet heel drastisch, maar geven toch te denken.
De verdeling van de kinderen volgt de normale verdeling erg netjes, zoals ook te zien is aan de QQ-plot.
De beschrijving zegt: “This data set lists the individual observations for 934 children in 205 families (…)”. Er zijn veel meer kinderen dan gezinnen in deze dataset. Dat wil zeggen dat er voor hetzelfde ouderpaar vaak meerdere kinderen in de dataset zijn opgenomen.
Er zijn dus groepjes van datapunten met dezelfde mid-ouder-lengte. Dat geeft een extra verklaring voor de observatie dat sommige punten in de puntenwolk verticaal uitlijnen: dat zijn kinderen van dezelfde ouders. Het zou ook kunnen verklaren dat de verdeling van de lengtes van de ouders ondanks het grote aantal waarnemingen niet zo netjes normaal verdeeld is.
Een consequentie is ook dat deze punten zeker niet onafhankelijk uit een bivariate kansverdeling getrokken zijn. Al met al kunnen we de standaard correlatietoets dus niet op deze gegevens toepassen.
–
De structuur van ouders_uniek is:
str(ouders_uniek)'data.frame': 205 obs. of 3 variables:
$ family: Factor w/ 205 levels "001","002","003",..: 1 2 3 4 5 6 7 8 9 10 ...
$ father: num 78.5 75.5 75 75 75 74 74 74 74.5 74 ...
$ mother: num 67 66.5 64 64 58.5 68 68 66.5 66 65.5 ...Er zijn 205 ouder-paren in de dataset opgenomen.
Code:
De gegevens zien er redelijk bivariaat normaal verdeeld uit. Er is niet duidelijk een verband te zien.
Je maakt de figuren zo:
::: {.cell}
```{.r .cell-code}
hist(ouders_uniek$father, main="vaders")
```
::: {.cell-output-display}
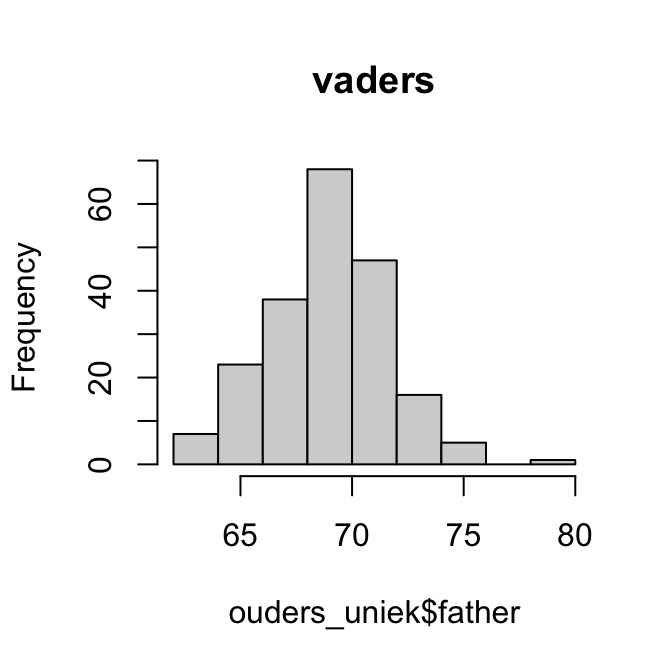{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
hist(ouders_uniek$mother, main="moeders")
```
::: {.cell-output-display}
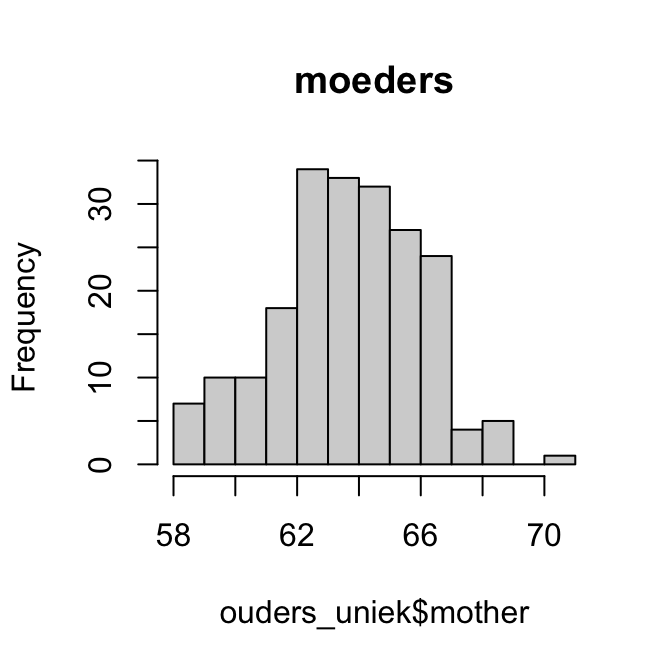{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
qqnorm(ouders_uniek$father, main="vaders")
qqline(ouders_uniek$father)
```
::: {.cell-output-display}
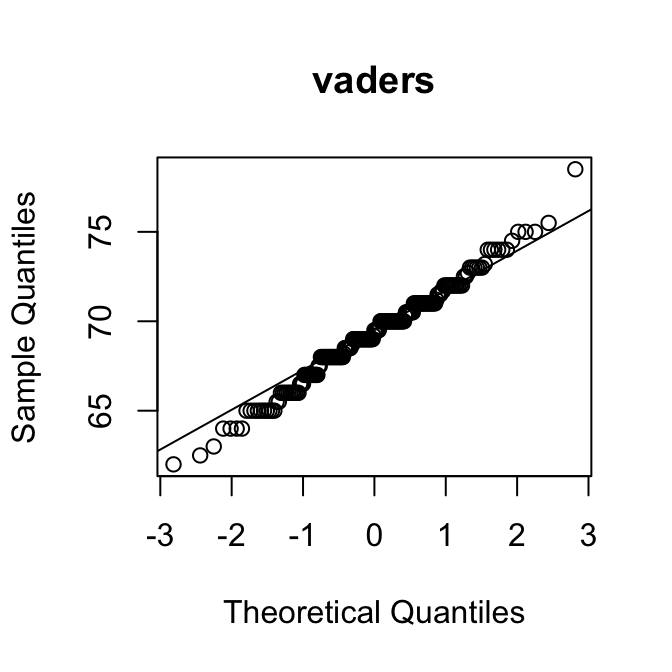{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
qqnorm(ouders_uniek$mother, main="moeders")
qqline(ouders_uniek$mother)
```
::: {.cell-output-display}
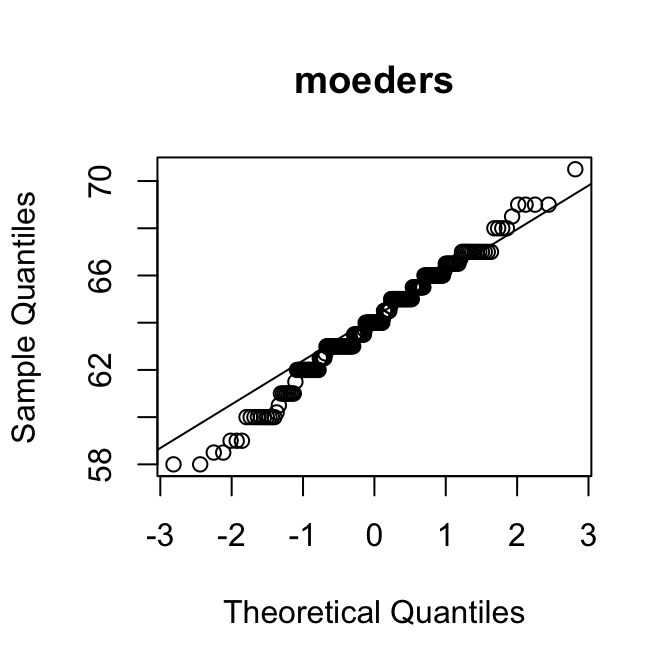{width=100%}
:::
:::Zowel de moeders als de vaders laten een verdeling zien
die wel iets wegheeft van een klokvorm,
maar niet heel netjes de normale verdeling volgt.
Het valt vooral op dat in beide QQ-plots
de Sample Quantiles voor kleine waarden kleiner zijn
dan verwacht op basis van de Theoretical Quantiles.
Dat geeft aan dat de verdelingen iets links-scheef zijn.
Het toepassen van de correlatie-test
is in dit geval dus dubieus.
Het zou beter zijn om een andere toets te gebruiken,
zoals Spearman's correlatie-toets,
maar die behandelen we in deze cursus niet.Hoewel we twijfelen of het bivariate model wel geschikt is, voeren we de toets toch uit:
cor.test(ouders_uniek$father, ouders_uniek$mother)
Pearson's product-moment correlation
data: ouders_uniek$father and ouders_uniek$mother
t = 1.3871, df = 203, p-value = 0.1669
alternative hypothesis: true correlation is not equal to 0
95 percent confidence interval:
-0.04067579 0.23086874
sample estimates:
cor
0.09689934 De \(P\)-waarde van 0,17 is verre van significant. De gegevens geven dus geen bewijs voor assorted mating.
Het betrouwbaarheidsinterval voor de ware correlatie in de populatie is \([-0{,}04; 0{,}23]\). Een correlatie van \(\rho \approx 0{,}2\) is dus nog niet uit te sluiten. Het is dus goed mogelijk dat een grotere steekproef wel een lichte correlatie aan het licht zou brengen.
Gebruik de regressielijn:
\(Y = 1{,}652448\times 10^2\textrm{\,mm} + (6{,}676867 \times 10^{-3}\textrm{\,mm/g}) \times 4000\textrm{\,g} = 192\textrm{\,mm}.\)
Gebruik de regressielijn:
\(Y = 1{,}652448\times 10^2\textrm{\,mm} + (6{,}676867 \times 10^{-3}\textrm{\,mm/g}) \times 500\textrm{\,g} = 169\textrm{\,mm}.\)
De regressielijn was bepaald voor pinguïns met een gewicht tussen \(2850\textrm{\,g}\) en \(4775\textrm{\,g}\). Bij onderdeel b hebben we de regressielijn toegepast op een veel kleiner gewicht van 500g. Dat is erg gevaarlijk: niemand kan weten of het lineaire model zo ver buiten dat domein nog van toepassing is.
In dat geval lijkt dat onwaarschijnlijk. Een pinguïn van 4000g heeft volgens de regressielijn gemiddeld een flipperlengte van 192mm. Het lijkt onwaarschijnlijk dat pinguïns die 8 keer lichter zijn, gemiddeld flippers hebben die met 169mm maar zo’n 12% korter zijn.
Een model toepassen buiten het domein waarvoor het bedoeld was, heet extrapoleren; we komen daar in Interpolatie en extrapolatie nog even op terug.
Data:
OD <- c( 31.0, 34.6, 40.0, 35.2, 57.5, 56.5, 57.0, 56.8)
drooggewicht <- c(24, 26, 32, 26, 42, 44, 44, 42)
monod <- data.frame(OD, drooggewicht)Plot van drooggewicht tegen OD:
Er is een mooi lineair verband; OD lijkt dus een erg goede voorspeller voor drooggewicht. Het is jammer dat verschillende punten dicht bij elkaar liggen. We hadden het verband beter kunnen controleren als ze beter verspreid waren.
Bereken de correlatiecoëfficiënt:
cor(monod$OD, monod$drooggewicht)[1] 0.9918988Een correlatiecoëfficiënt van 0,99 is heel erg hoog; dit is een erg sterk lineair verband.
We runnen eerst de lm() functie, en vragen dan de coëfficiënten op met de functie summary().
resultaatLM <- lm(drooggewicht ~ OD, data = monod)
summary(resultaatLM)$coefficients Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.8342606 1.83751520 0.4540156 6.657779e-01
OD 0.7415245 0.03876953 19.1264753 1.321125e-06Lees de coëfficiënten af; dat geeft de volgende regressielijn:
\(\textrm{drooggewicht} = 0{,}834\textrm{\,mg/L} + (0{,}742\textrm{\,mg/L}) \cdot \textrm{OD}.\)
Voor de volledigheid kunnen we de regressielijn aan de plot toevoegen:
Vul de waarde in de regressielijn in:
a <- 0.8342606
b <- 0.7415245
OD_waarde <- 45.5
( drooggewicht_voorspelling <- a + b*OD_waarde )[1] 34.57363Op zich kunnen we de regressielijn gebruiken om een voorspelling te doen:
OD_waarde_2 <- 100.0
( drooggewicht_voorspelling_2 <- a + b*OD_waarde_2 )[1] 74.98671Maar de OD van 100,0 ligt erg ver buiten het domein van de gegevens waaraan de regressielijn was gefit. Als je zo ver extrapoleert is er geen enkele garantie dat de regressielijn nog werkt als proxy voor het drooggewicht. Het is dus heel onverstandig om op deze voorspelling te vertrouwen.
Bekijk de dataset:
str(anscombe)'data.frame': 11 obs. of 8 variables:
$ x1: num 10 8 13 9 11 14 6 4 12 7 ...
$ x2: num 10 8 13 9 11 14 6 4 12 7 ...
$ x3: num 10 8 13 9 11 14 6 4 12 7 ...
$ x4: num 8 8 8 8 8 8 8 19 8 8 ...
$ y1: num 8.04 6.95 7.58 8.81 8.33 ...
$ y2: num 9.14 8.14 8.74 8.77 9.26 8.1 6.13 3.1 9.13 7.26 ...
$ y3: num 7.46 6.77 12.74 7.11 7.81 ...
$ y4: num 6.58 5.76 7.71 8.84 8.47 7.04 5.25 12.5 5.56 7.91 ...De kengetallen voor x1 en y1:
mean(anscombe$x1)[1] 9mean(anscombe$y1)[1] 7.500909sd(anscombe$x1)[1] 3.316625sd(anscombe$y1)[1] 2.031568cor(anscombe$x1, anscombe$y1)[1] 0.8164205resultaatLM1 <- lm(y1 ~ x1, anscombe)
summary(resultaatLM1)$coefficients Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0000909 1.1247468 2.667348 0.025734051
x1 0.5000909 0.1179055 4.241455 0.002169629De kengetallen voor x2 en y2:
mean(anscombe$x2)[1] 9mean(anscombe$y2)[1] 7.500909sd(anscombe$x2)[1] 3.316625sd(anscombe$y2)[1] 2.031657cor(anscombe$x2, anscombe$y2)[1] 0.8162365resultaatLM2 <- lm(y2 ~ x2, anscombe)
summary(resultaatLM2)$coefficients Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.000909 1.1253024 2.666758 0.025758941
x2 0.500000 0.1179637 4.238590 0.002178816De kengetallen voor x3 en y3:
mean(anscombe$x3)[1] 9mean(anscombe$y3)[1] 7.5sd(anscombe$x3)[1] 3.316625sd(anscombe$y3)[1] 2.030424cor(anscombe$x3, anscombe$y3)[1] 0.8162867resultaatLM3 <- lm(y3 ~ x3, anscombe)
summary(resultaatLM3)$coefficients Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0024545 1.1244812 2.670080 0.025619109
x3 0.4997273 0.1178777 4.239372 0.002176305De kengetallen voor x4 en y4:
mean(anscombe$x4)[1] 9mean(anscombe$y4)[1] 7.500909sd(anscombe$x4)[1] 3.316625sd(anscombe$y4)[1] 2.030579cor(anscombe$x4, anscombe$y4)[1] 0.8165214resultaatLM4 <- lm(y4 ~ x4, anscombe)
summary(resultaatLM4)$coefficients Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0017273 1.1239211 2.670763 0.025590425
x4 0.4999091 0.1178189 4.243028 0.002164602Voor alle vier de datasets zijn de verschillende kengetallen voor ligging, spreiding, correlatie én regressie nagenoeg hetzelfde.
Op basis van deze uitgebreide set kengetallen zou je geneigd zijn te concluderen dat de datasets erg op elkaar lijken.
Alle plotjes:
::: {.cell}
```{.r .cell-code}
plot(anscombe$x1, anscombe$y1)
abline(resultaatLM1, col = "darkorange")
```
::: {.cell-output-display}
{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
plot(anscombe$x2, anscombe$y2)
abline(resultaatLM2, col = "darkorange")
```
::: {.cell-output-display}
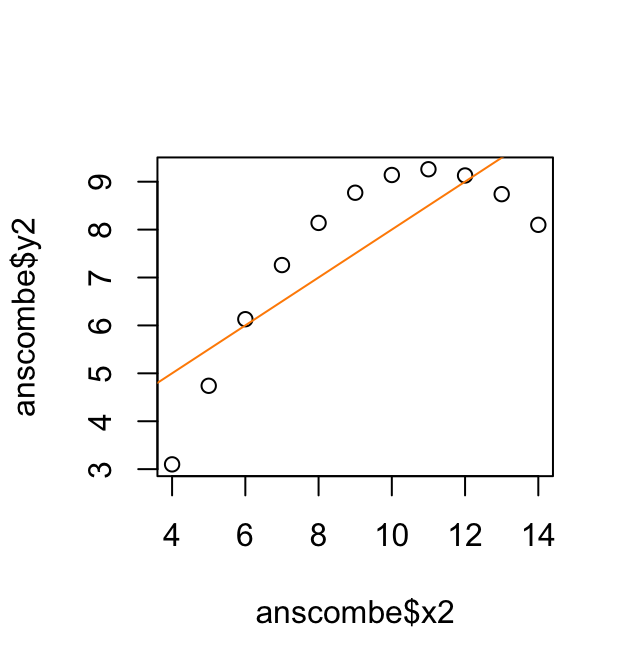{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
plot(anscombe$x3, anscombe$y3)
abline(resultaatLM3, col = "darkorange")
```
::: {.cell-output-display}
{width=100%}
:::
:::
::: {.cell}
```{.r .cell-code}
plot(anscombe$x4, anscombe$y4)
abline(resultaatLM4, col = "darkorange")
```
::: {.cell-output-display}
{width=100%}
:::
:::Hoewel de kengetallen van de vier datasets
vrijwel identiek zijn,
vertellen de plotjes allemaal een heel ander verhaal.
De eerste dataset laat een lineair verband zien.
In de tweede dataset is het verband duidelijk niet lineair;
het ziet er uit als een parabool.
De gegevens in de derde dataset hebben een lineair verband,
maar alle kengetallen en coëfficiënten worden sterk verstoord
door een uitbijter. Tot slot wordt in de vierde dataset
de volledige correlatie, regressie, en standaarddeviatie
bepaald door één enkel punt.
Alleen bij de eerste dataset zijn de correlatiecoëfficiënt
en de regressielijn betekenisvol.De correlatiecoëfficiënt voor de Chinstrap-pinguïns is sterk positief, zoals de wetenschappers verwacht hadden. Je kunt dit direct zien aan de puntenwolk en de oranje regressielijn.
De correlatiecoëfficiënt voor de Stubbybeak-pinguïns is ook positief,
Het is een vreemd fenomeen: voor beide soorten is het verband tussen snavellengte en flipperlengte positief, maar als beide datasets worden samengevoegd vinden we een negatieve correlatie.
Dit fenomeen heet Simpson’s paradox. Aan het plaatje kun je zien wat de paradox veroorzaakt. Binnen beide groepen is de correlatie positief, maar de gemiddelden van de groepen laten juist een negatieve correlatie zien: Stubbybeak heeft gemiddeld langere flippers, maar een kortere snavel dan Chinstrap. De zwarte regressielijn van de gehele populatie laat zich daardoor naar beneden trekken.
Onder a t/m d voer je het volgende script uit:
meetgegevens <- data.frame(
substraatconcentratie = c(0.1, 0.2, 0.5, 1.0, 2.0, 5.0, 10.0),
reactiesnelheid = c(2.3, 4.1, 8.5, 13.2, 18.4, 22.5, 24.0)
)# Definieer de Michaelis-Menten-curve
michaelis_menten <- function(S, v_max, K_M) {
v_max * S / (K_M + S)
}
# Pas niet-lineaire regressie toe
fit <- nls(
reactiesnelheid ~ michaelis_menten(substraatconcentratie, v_max, K_M),
data = meetgegevens,
start = list(v_max = 25, K_M = 1) # Startwaarden
)
summary(fit)$coefficients Estimate Std. Error t value Pr(>|t|)
v_max 26.91670 0.43182619 62.33226 2.011605e-08
K_M 1.02837 0.05451145 18.86521 7.709198e-06De coëfficiënten liggen inderdaad in de buurt van de getallen die we vooraf hadden ingeschat.
Plot data met de fit:
v_max <- 26.91670
K_M <- 1.02837
S_waarden <- seq(0, 10, 0.5)
# de gefitte MM-curve:
reactiesnelheid_model <- michaelis_menten(S_waarden, v_max, K_M)
# plot de lijn volgens het model
plot(
S_waarden, reactiesnelheid_model,
type = "l", col = "darkorange", lwd = 3,
xlab = "substraat",
ylab = "reactiesnelheid")
# voeg de datapunten eraan toe
points(
meetgegevens$substraatconcentratie,
meetgegevens$reactiesnelheid,
pch = 21,
bg = "gray",
cex = 1.5
)De gegevens liggen netjes langs het model. Het model lijkt dus erg geschikt om deze gegevens te beschrijven.